- Cinetocoro
-
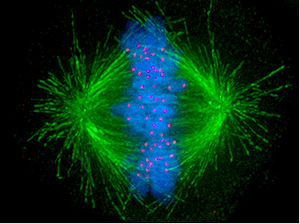 Imagen de una célula humana en mitosis, en la que los microtúbulos se muestran en verde (formando el huso mitótico), los cromosomas en azul en el ecuador del huso y los cinetocoros en rojo.
Imagen de una célula humana en mitosis, en la que los microtúbulos se muestran en verde (formando el huso mitótico), los cromosomas en azul en el ecuador del huso y los cinetocoros en rojo.
El cinetocoro es una estructura proteica situada sobre los cromosomas. Sobre esta estructura se anclan los microtúbulos (MTs) del huso mitótico durante los procesos de división celular (meiosis y mitosis). El cinetocoro está localizado en una zona específica del cromosoma, el centrómero. En vertebrados y levaduras los cinetocoros son estructuras discretas y únicas en cada cromosoma, pero existen organismos (como C. elegans) que presentan cinetocoros difusos a lo largo de los brazos cromosómicos: son los denominados cromosomas holocéntricos.[1]
Los cinetocoros inician, controlan y supervisan los llamativos movimientos de los cromosomas durante la división celular. En cuanto a su estructura, los cinetocoros de las células animales pueden subdividirse en dos regiones:
- el cinetocoro interno se organiza normalmente sobre secuencias de ADN altamente repetido (el ADN satélite) y se ensambla en una forma especializada de cromatina que persiste a través del ciclo celular.
- el cinetocoro externo es una estructura proteica con muchos componentes dinámicos que se ensambla y funciona sólo durante la división celular.
Las funciones del cinetocoro incluyen el anclaje de los cromosomas a los MTs del huso mitótico, la verificación de esos anclajes, la activación del checkpoint de mitosis (un mecanismo de control que retrasa la salida de mitosis si se detectan fallos) y la participación en la generación de las fuerzas que propulsan los movimientos cromosómicos durante la división celular.[2]
Los microtúbulos son polímeros metaestables de tubulina-α y β que alternan entre fases de crecimiento y despolimerización, un fenómeno que se conoce como "inestabilidad dinámica".[3] La naturaleza enormemente dinámica del comportamiento de los MTs está integrada con la función de los cinetocoros para mover y segregar los cromosomas.
Contenido
Estructura del cinetocoro en animales
El cinetocoro está compuesto de distintas capas, que se observaron inicialmente por métodos convencionales de fijación y teñido de microscopía electrónica[4] [5] (revisado por C. Rieder en 1982[6] ), y más recientemente por congelación rápida y sustitución.[7]
 Estructura y componentes de los cinetocoros en vertebrados. Basado en Maiato et al. (2004).[2]
Estructura y componentes de los cinetocoros en vertebrados. Basado en Maiato et al. (2004).[2]
La capa más profunda del cinetocoro es la placa interna, que se organiza sobre una estructura de cromatina que contiene nucleosomas que presentan una histona especializada (CENP-A, que sustituye a la histona H3 en esta zona), proteínas auxiliares y ADN. La organización de este ADN es uno de los aspectos más desconocidos del cinetocoro de vertebrados. La placa interna aparece como un dominio de heterocromatina discreto a través de todo el ciclo celular. Por fuera de ésta aparece la placa externa, compuesta sobre todo de proteínas. Esta estructura se forma en la superficie de los cromosomas en el momento de la ruptura de la envoltura nuclear.[4] La placa externa de los cinetocoros de vertebrados tiene alrededor de 20 sitios de anclaje para extremos (+) de microtúbulos (denominados kMTs, por kinetochore MTs), mientras que la placa externa de los cinetocoros de Saccharomyces cerevisiae tiene un solo sitio de anclaje. La zona más externa del cinetocoro forma una corona fibrosa que puede visualizarse por microscopía convencional, pero sólo en ausencia de MTs. Esta corona esta formada por una red dinámica de proteínas residentes y temporales que están implicadas en el checkpoint de mitosis, en el anclaje de MTs y en la regulación del comportamiento de éstos.
Durante mitosis, cada una de las dos cromátidas hermanas que forman el cromosoma completo presenta su propio cinetocoro. Los cinetocoros hermanos distintos se observan por primera vez al final de la fase G2 en células cultivadas de mamíferos.[8] Estos cinetocoros tempranos presentan una estructura laminar madura antes de la ruptura de la envoltura nuclear (revisado por Pluta y colaboradores en 1995[9] ). La ruta molecular del ensamblaje de los cinetocoros de los eucariotas superiores se ha estudiado utilizando knockouts de genes en ratones y en células de pollo en cultivo, así como técnicas de ARN interferente (RNAi) en C. elegans, Drosophila y células humanas. Sin embargo, ninguna ruta lineal simple puede describir los datos obtenidos.
La primera proteína que se ensambla en el cinetocoro es CENP-A (Cse4 en Saccharomyces cerevisiae). Esta proteína es una isoforma especializada de la histona H3.[10] CENP-A es necesaria para la incorporación de las proteínas del cinetocoro interno CENP-C, CENP-H y CENP-I/MIS6.[11] [12] [13] [14] [15] Las posiciones relativas de estas proteínas en la ruta dependiente de CENP-A no están completamente claras. Por ejemplo, la localización de CENP-C requiere CENP-H en células de pollo, pero es independiente de CENP-I/MIS6 en células humanas. En C. elegans y en metazoos, la incorporación de muchas proteínas del cinetocoro externo depende en último término de CENP-A.
Los componentes del cinetocoro pueden agruparse en función de su localización a través del ciclo celular. Los componentes constitutivos, como CENP-A, CENP-C, CENP-H y CENP-I, están unidos a la cromatina asociada al cinetocoro durante todo el ciclo, mientras que otros componentes sólo se asocian al cinetocoro al comenzar la profase.
Las proteínas del cinetocoro también pueden agruparse en función de si su concentración cinetocórica permanece constante o varía durante mitosis, y si se reciclan de forma lenta (son estables) o rápida (dinámicas) en sus sitios de unión en los cinetocoros.
- Las proteínas que permanecen en niveles prácticamente estables desde profase hasta anafase tardía incluyen los componentes constitutivos de la placa interna y los componentes estables del cinetocoro externo, tales como el complejo Ndc80,[16] [17] las proteínas KNL/KBP (kinetochore-null/KNL-binding protein),[18] proteínas MIS[18] y CENP-F.[19] [20] Conjuntamente con los componentes constitutivos, estas proteínas parecen formar el núcleo central de las estructuras de las placas interna y externa del cinetocoro.
- Los componentes dinámicos cuya concentración en los cinetocoros cambia durante mitosis incluyen los motores moleculares CENP-E y dineína (además de sus componentes diana ZW10 y ROD), y las proteínas del checkpoint de mitosis (como Mad1, Mad2, BubR1 y Cdc20). Estas proteínas se ensamblan en el cinetocoro en altas concentraciones en ausencia de microtúbulos y su concentración disminuye a medida que aumenta el número de microtúbulos anclados al cinetocoro.[21] En metafase, los niveles de CENP-E, Bub3 y Bub1 disminuyen de 3 a 4 veces con relación a los presentes en cinetocoros no anclados, mientras que los niveles de dineína/dinactina, Mad1, Mad2 y BubR1 caen >10-100 veces.[21] [22] [23] [24]
- Mientras que las proteínas del checkpoint de mitosis presentes en la placa externa del cinetocoro disminuyen su concentración cuando los MTs se anclan,[24] otros componentes como EB1, APC y las proteínas de la ruta Ran (RanGap1 y RanBP2) se asocian con los cinetocoros sólo cuando los microtúbulos se anclan.[25] [26] [27] [28] Esto podría ser parte de un mecanismo en el cinetocoro que reconoce el extremo (+) de los MTs, asegura que están anclados correctamente y regula su comportamiento dinámico mientras permanecen anclados.
Un estudio del 2010 utiliza un método complejo (denominado proteómica combinatoria con clasificadores múltiples o MCCP por las siglas en inglés de multiclassifier combinatorial proteomics) para analizar la composición proteica de los cromosomas completos de vertebrados, incluyendo el cinetocoro.[29] Aunque este estudio no incluye un proceso bioquímico de enriquecimiento en cinetocoros, los datos obtenidos incluyen todos los sub-complejos centroméricos, con péptidos procedentes de 125 proteínas centroméricas conocidas. Según este estudio, aún se desconocen del orden de un centenar de proteínas asociadas al cinetocoro, lo que dobla la complejidad de la estructura conocida durante la mitosis, confirmando que el cinetocoro es una de las sub-estructuras más complejas de la célula.
Función de los cinetocoros
El número de microtúbulos que se unen a un cinetocoro es variable: en Saccharomyces cerevisiae se une al cinetocoro tan sólo un microtúbulo, mientras que en mamíferos superiores a cada cinetocoro se unen entre 15 y 35 microtúbulos.[30] Sin embargo, no todos los microtúbulos del huso alcanzan los cinetocoros. Hay microtúbulos que se extienden desde un centrosoma al otro (de los que depende la longitud del huso) y otros más cortos que están interdigitados entre los microtúbulos largos. El profesor B. Nicklas, en la Universidad de Carolina del Norte, demostró que si se rompe la unión entre los microtúbulos y los cinetocoros mediante un rayo láser, las cromátidas no pueden moverse, produciéndose una distribución anormal de los cromosomas.[31] Estos experimentos demostraron también que el cinetocoro tiene polaridad y que su interacción con los microtúbulos de uno u otro centrosoma dependerá de su orientación. Esta especificidad garantiza que solamente una de las cromátidas se mueva hacia cada lado del huso, asegurando la distribución adecuada del material genético. Una de las funciones básicas del cinetocoro es, por tanto, el anclaje a los microtúbulos del huso, que es fundamental para realizar una correcta segregación de cromátidas. Si el anclaje se produce de forma incorrecta, se pueden producir errores que generen situaciones de aneuploidía, con drásticas consecuencias para la célula. Para evitarlo, existen mecanismos de detección y corrección de errores (como el checkpoint de mitosis), cuyos componentes también residen en los cinetocoros. El desplazamiento de una cromátida hacia el centrosoma se produce fundamentalmente por despolimerización de los microtúbulos en el sitio de unión con el cinetocoro. Dichos desplazamientos precisan además la generación de fuerzas en las que intervienen motores moleculares localizados asimismo en los cinetocoros.
Anclaje de los cromosomas a los MTs del huso mitótico
* Captura de MTs
 Los cromosomas se enganchan al huso mitótico a través de los cinetocoros de ambas cromátidas hermanas, en una orientación bipolar.
Los cromosomas se enganchan al huso mitótico a través de los cinetocoros de ambas cromátidas hermanas, en una orientación bipolar.
Durante la fase de síntesis (fase S) del ciclo celular, el centrosoma comienza a duplicarse. Justo al inicio de mitosis, ambos centriolos de cada centrosoma alcanzan su longitud máxima, los centrosomas reclutan material adicional y su capacidad de nucleación de microtúbulos aumenta. A medida que progresa la mitosis, ambos centrosomas se separan para establecer el huso mitótico.[32] De esta forma, el huso de una célula mitótica tiene dos polos que emanan microtúbulos. Los microtúbulos son largos filamentos protéicos con dos extremos asimétricos, un extremo "menos" (-) relativamente estable cercano al centrosoma, y un extremo "más" (+) que sufre fases alternadas de crecimiento-retroceso y que explora el centro celular. En este proceso de búsqueda, un microtúbulo puede localizar y capturar un cromosoma.[33] [34] Los microtúbulos que localizan un cinetocoro se estabilizan, mientras que aquellos que no lo encuentran se despolimerizan rápidamente.[35] Como los cromosomas presentan dos cinetocoros asociados espalda-con-espalda (uno en cada cromátida hermana), cuando uno de ellos se engancha a los microtúbulos generado por uno de los polos celulares, el cinetocoro de la cromátida hermana queda expuesto hacia el otro polo celular, por lo que en la mayor parte de los casos el segundo cinetocoro se asocia a los microtúbulos del polo opuesto,[36] de manera que los cromosomas quedan « bi-orientados», una configuración fundamental (también denominada anfitélica) para asegurar que la segregación tendrá lugar de manera correcta cuando la célula se divida.[37] [38]

En el momento en que un solo microtúbulo se ancla a un cinetocoro, se inicia un rápido movimiento del cromosoma asociado en dirección al polo del que procede dicho microtúbulo. Este movimiento está probablemente mediado por la actividad motora dirigida hacia el extremo (-) de la proteína motora dineína citoplásmica,[39] [40] que se encuentra muy concentrada en los cinetocoros sin anclar (revisado por Banks y Heald en 2001[41] ). El movimiento hacia el polo se ralentiza a medida que los cinetocoros adquieren kMTs (MTs anclados a cinetocoros) y el movimiento pasa a ser dirigido por cambios en la longitud de los kMTs. La dineína se libera de los cinetocoros a medida que éstos adquieren kMTs[21] y, en células en cultivo de mamíferos, es necesaria para la inactivación del checkpoint de mitosis, pero no para el alineamiento cromosómico en el ecuador del huso, la adquisición de kMTs o la anafase A durante la segregación cromosómica.[42] En plantas superiores o en levaduras no hay evidencia de existencia de dineína, pero otras kinesinas dirigidas hacia el extremo (-) podrían compensar la falta de dineína.
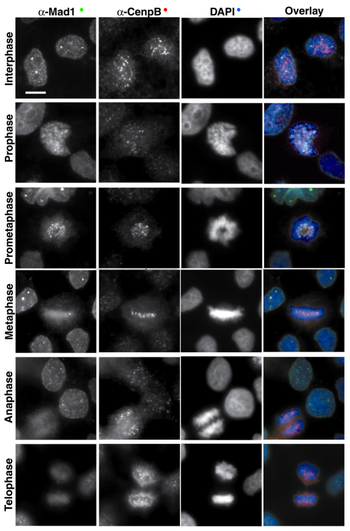
 Células en metafase con niveles bajos de CENP-E mediante RNAi, que muestran cromosomas no alineados en la placa metafásica (flechas). Estos cromosomas presentan marcaje para las proteínas del checkpoint de mitosis Mad1/Mad2. Hec1 y CENP-B marcan la región centromérica (el cinetocoro), y DAPI es una tinción específica para el ADN.
Células en metafase con niveles bajos de CENP-E mediante RNAi, que muestran cromosomas no alineados en la placa metafásica (flechas). Estos cromosomas presentan marcaje para las proteínas del checkpoint de mitosis Mad1/Mad2. Hec1 y CENP-B marcan la región centromérica (el cinetocoro), y DAPI es una tinción específica para el ADN.
Otra proteína motora implicada en la captación inicial de MTs es CENP-E; ésta es una kinesina de gran tamaño que se asocia con la corona fibrosa de los cinetocoros de mamíferos desde prometafase hasta anafase.[43] En células con niveles bajos de CENP-E, los cromosomas carecen de esta proteína en sus cinetocoros, que a menudo presentan defectos en su capacidad de alinearse. En este caso, algunos cromosomas permanecen crónicamente mono-orientados (anclados a un único polo), incluso aunque la mayoría se congreguen correctamente en la placa metafásica.[44]
En general se acepta ampliamente que la fibra de kMTs (el haz de microtúbulos unidos al cinetocoro) se forma originalmente por la captura de MTs que se polimerizaron en los centrosomas y polos del huso en células de mamíferos en cultivo.[33] Sin embargo, la polimerización de MTs directamente en los cinetocoros podría también contribuir de forma importante.[2] La forma en la cual la región del cinetocoro/centrómero inicia la formación de fibras de kMTs y con qué frecuencia ocurre son cuestiones importantes, dado que este mecanismo puede contribuir no sólo a la formación inicial de kMTs, sino también a cómo los cinetocoros corrigen errores en el anclaje de MTs y regulan el movimiento a lo largo de los kMTs.
* Papel del complejo Ndc80
Los MTs asociados a cinetocoros presentan unas características especiales: en comparación a los MTs no anclados, los kMTs son mucho más resistentes a la despolimerización inducida por el frío, las altas presiones hidrostáticas o la exposición al calcio.[45] Además, los kMTs se reciclan mucho más despacio que los MTs astrales y los MTs del huso que tienen extremos (+) libres, y si los kMTs se separan de los cinetocoros mediante rayo láser, se despolimerizan rápidamente.[31]
Una vez establecido que dineína y CENP-E no son esenciales para la formación de los kMTs, las moléculas responsables de la estabilización de éstos debían de ser otras. Estudios genéticos pioneros en levaduras revelaron la importancia del complejo Ndc80 en el anclaje de los kMTs.[16] [46] [47] [48] En Saccharomyces cerevisiae, el complejo Ndc80 tiene cuatro componentes: Ndc80p, Nuf2p, Spc24p y Spc25p. Los mutantes que carecen de alguno de los componentes de este complejo presentan pérdida de la conexión cinetocoro-microtúbulo sin mostrar una pérdida completa de la estructura del cinetocoro.[16] [46] Sin embargo, los mutantes en los que se ha perdido la estructura del cinetocoro (como los mutantes para Ndc10 en levaduras[49] ) presentan deficiencias tanto en la conexión con los microtúbulos como en la capacidad de respuesta del checkpoint de mitosis, posiblemente porque los cinetocoros funcionan como una plataforma en la que se organizan los componentes de la respuesta.
El complejo Ndc80 está muy conservado y se ha identificado en S. pombe, C. elegans, Xenopus, pollos y humanos.[16] [46] [50] [51] [17] [52] [53] Estudios realizados con Hec1 (por highly expressed in cancer cells 1), el homólogo de Ndc80p en humanos, han mostrado que es importante para la correcta congregación cromosómica y la progresión a través de mitosis, y que interacciona con componentes de los complejos de cohesinas y condensinas.[54]
Diferentes laboratorios han mostrado que el complejo Ndc80 es crucial para la estabilización de los anclajes cinetocoro-microtúbulo, necesarios para mantener las tensiones centroméricas implicadas en el establecimiento del alineamiento cromosómico correcto en eucariotas superiores.[51] [17] [52] [53] Las células en las que se ha eliminado la función de Ndc80 (mediante RNAi, knockout de genes, o microinyección de anticuerpos) tienen husos mitóticos anormalmente largos, pérdida de tensión entre cinetocoros hermanos, cromosomas que no pueden congregarse en la placa metafásica y pocos o ningún kMT asociados.
Aunque existe alguna evidencia in vitro de que heterodímeros Ndc80p-Nuf2p pueden unirse a microtúbulos,[55] no está claro que in vivo la conexión entre el complejo Ndc80 y los MTs sea directa. En levaduras, esta conexión requiere la presencia del complejo Dam1-DASH-DDD. Algunos miembros de este complejo se unen directamente a los MTs, mientras que otros se unen al complejo Ndc80.[47] [48] [56] Por tanto, el complejo Dam1-DASH-DDD podría ser un adaptador esencial entre los cinetocoros y los microtúbulos. Sin embargo, en animales no se ha encontrado un complejo equivalente, y esta cuestión permanece bajo intensa investigación.
Verificación de los anclajes al huso
Cuando una célula entra en mitosis, duplica toda su información genética, en el proceso denominado replicación del ADN. Al final de este proceso, cada cromosoma consta de dos cromátidas hermanas, que son dos moléculas de ADN completas e idénticas. Ambas cromátidas permanecen unidas mediante complejos cohesinas hasta anafase, cuando se produce la segregación cromosómica. Para que ésta se produzca correctamente, cada célula hija debe recibir un juego completo de cromátidas, lo cual quiere decir que cada cromátida hermana (a través de su cinetocoro) debe anclarse a MTs procedentes de polos opuestos del huso mitótico. Esta configuración se denomina anfitélica o "bi-orientación".
Sin embargo, durante el proceso de anclaje se producen también configuraciones incorrectas:[57]
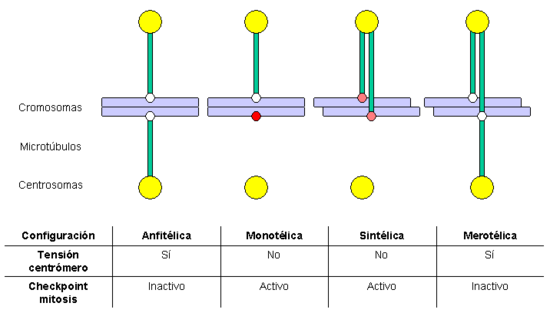 Esquema de las diferentes configuraciones de anclaje de los cromosomas al huso mitótico.[2]
Esquema de las diferentes configuraciones de anclaje de los cromosomas al huso mitótico.[2]
- monotélica: sólo una de las dos cromátidas se ancla a MTs, el segundo cinetocoro no se ancla; por tanto, no se genera tensión centromérica, con lo cual se activa el checkpoint de mitosis, que retrasa la entrada en anafase y da tiempo a la célula a corregir el error. Si no se corrigiera, la cromátida no anclada podría incluirse al azar en cualquiera de las dos células hijas, lo que generaría aneuploidía: una célula hija tendría cromosomas de más y la otra carecería de algún cromosoma.
- sintélica: las dos cromátidas hermanas se anclan a MTs que proceden del mismo polo; esta situación tampoco genera tensión centromérica, con lo cual se activa el checkpoint de mitosis. Si no se corrige, ambas cromátidas se dirigirán a la misma célula hija, generando una situación de aneuploidía.
- merotélica: al menos una cromátida se ancla simultáneamente a MTs generados por ambos polos. Esta situación genera tensión centromérica, por lo que no activa el checkpoint de mitosis. Si no se corrige, la cromátida unida a ambos polos simultáneamente permanecerá como un cromosoma retrasado en anafase, y finalmente se romperá en dos fragmentos, que se repartirán entre las células hijas, generando aneuploidía.
Tanto la configuración monotélica como la sintélica generan tensión centromérica y son detectadas por el checkpoint de mitosis, pero la configuración merotélica no se detecta por este mecanismo de control. Sin embargo, la mayor parte de estos errores son detectados y corregidos antes de que la célula entre en anafase.[57] Un factor clave en la corrección de los errores de anclaje es el complejo pasajero de los cromosomas (chromosomal passenger complex), que incluye la proteína kinasa Aurora B, su diana y subunidad activadora INCENP y otras dos subunidades, Survivina y Borealina/Dasra B (véase la revisión de Adams y colaboradores en 2001[58] ). En células en las que se ha eliminado la función de este complejo mediante mutantes dominantes-negativos, RNAi, microinyección de anticuerpos o utilización de drogas selectivas, se acumulan errores en el anclaje de los cromosomas. Muchos estudios han mostrado que Aurora B es necesaria para desestabilizar los anclajes cinetocoro-microtúbulo incorrectas, de manera que se favorezca la generación de conexiones anfitélicas. El homólogo de Aurora B en levaduras (Ipl1p) fosforila algunas proteínas del cinetocoro, como la proteína constitutiva Ndc10p y miembros de los complejos Ndc80 y Dam1-DASH-DDD.[59] La fosforilación de los componentes del complejo Ndc80 produce la desestabilización de los anclajes de los kMTs. Se ha propuesto que la localización de Aurora B es importante para su función: al encontrarse en la zona interna del cinetocoro (en la heterocromatina centromérica), cuando se establece tensión centromérica los cinetocoros hermanos se alejan, y Aurora B no puede alcanzar sus sustratos, de manera que los kMTs se estabilizan. Es interesante notar que Aurora B se encuentra sobreexpresada frecuentemente en varios tipos de tumores y es en la actualidad una diana para el desarrollo de drogas anticancerosas.[60]
Activación del checkpoint de mitosis
El mecanismo que detecta que se ha formado correctamente un huso mitótico, que todos los cromosomas están asociados a dicho huso de manera bipolar, y que todos ellos se encuentran alineados en la placa metafásica es el denominado checkpoint de mitosis o también punto de control del ensamblaje del huso, abreviado SAC por sus siglas en inglés (Spindle Assembly Checkpoint). Cuando alguno de los cromosomas, por alguna razón, se retrasa durante el proceso de alineamiento, esta maquinaria produce una parada temporal de la progresión en el ciclo celular: la célula se detiene, dando tiempo a los mecanismos de reparación a resolver el problema detectado. Si pasado un tiempo, el problema no se ha corregido, la célula será abocada a un proceso de muerte celular, un mecanismo de seguridad para evitar que se produzca una situación de aneuploidía, generalmente con consecuencias graves para el organismo.
Mientras que las proteínas centroméricas estructurales (como CENP-B), mantienen una localización estable a lo largo de toda la mitosis, incluida telofase, los componentes del checkpoint de mitosis se ensamblan en el cinetocoro en altas concentraciones en ausencia de microtúbulos y su concentración disminuye a medida que aumenta el número de microtúbulos anclados al cinetocoro.[21] En metafase, los niveles de CENP-E, Bub3 y Bub1 disminuyen de 3 a 4 veces con relación a los presentes en cinetocoros no anclados, mientras que los niveles de dineína/dinactina, Mad1, Mad2 y BubR1 caen >10-100 veces.[21] [22] [23] [24] Por tanto en metafase, cuando todos los cromosomas están congregados en la placa metafásica, se liberan las proteínas del checkpoint. La desaparición de las proteínas del checkpoint de los cinetocoros marca el momento en que los cromosomas han alcanzado la placa metafásica y se encuentran en tensión bipolar. Es entonces cuando las proteínas del checkpoint que se unen e inhiben Cdc20 (Mad1-Mad2 y BubR1), lo liberan, permitiendo la activación del APC/CCdc20, que dispara la separación de las cromátidas hermanas y consecuentemente el inicio de la anafase.
Varios estudios indican que el complejo Ndc80 interviene en la regulación de la asociación estable de Mad1-Mad2 y dineína con los cinetocoros.[17] [52] [53] Sin embargo, las proteínas asociadas con el cinetocoro CENP-A, CENP-C, CENP-E, CENP-H y BubR1 son independientes de Ndc80/Hec1. La prolongada parada en prometafase que se observa en células con niveles reducidos de Ndc80/Hec1 depende de Mad2, a pesar del hecho de que en estas células muestran niveles de Mad1, Mad2 y dineína en sus cinetocoros que son menores del 10-15% a los observados en cinetocoros no anclados. Sin embargo, si además de Ndc80/Hec1 se disminuyen los niveles de Nuf2, Mad1 y Mad2 desaparecen completamente de los cinetocoros y el checkpoint se inactiva.[61]
Las proteínas denominadas Shugoshinas (MEI-S332 en Drosophila melanogaster[62] ) son un tipo de proteínas asociadas con los centrómeros que son esenciales para mantener las cohesinas unidas a los centrómeros hasta anafase. La proteína homóloga en humanos, hsSgo1, se asocia con los centrómeros durante profase y desaparece al inicio de la anafase.[63] Cuando se disminuyen los niveles de Shugoshina mediante RNAi en células HeLa, las cohesinas no se pueden mantener en los centrómeros durante mitosis, y en consecuencia, las cromátidas hermanas se separan asincrónicamente antes de que se inicie la anafase, lo que dispara una parada mitótica prolongada.
Por otro lado, el grupo de Dasso y colaboradores encontraron que las proteínas del sistema Ran: RanGAP1 (una proteína activadora de GTPasas que estimula la conversión de Ran-GTP en Ran-GDP) y la proteína de unión a Ran denominada RanBP2/Nup358, pueden detectarse en los cinetocoros durante mitosis.[64] Estas proteínas se encuentran en los poros nucleares durante interfase e intervienen en el transporte nucleo-citoplásmico. La localización cinetocórica de estas proteínas parece ser significativa funcionalmente, porque una variedad de tratamientos que elevan los niveles de Ran-GTP inhiben la liberación de las proteínas Bub1, Bub3, Mad2 y CENP-E de los cinetocoros.[65]
Curiosamente, Orc2 (una proteína del complejo de reconocimiento de origen -ORC por sus siglas en inglés- implicado en la iniciación de la replicación del ADN durante la fase S) también se localiza en los cinetocoros durante mitosis en células humanas;[66] en concordancia con esta localización, algunos estudios indican que Orc2 en levaduras está implicada en la cohesión de cromátidas hermanas, y su eliminación celular provoca la activación del checkpoint de mitosis.[67] También se ha detectado que otros componentes del complejo ORC (como orc5 en S. pombe) intervienen en cohesión.[68] Sin embargo, la ruta molecular en la que intervienen las proteínas ORC parece ser aditiva a la ruta de las cohesinas y se desconoce en su mayor parte.
Generación de las fuerzas que propulsan los movimientos cromosómicos
Véase también: MicrotúbuloLa mayor parte de los movimientos cromosómicos en relación con los polos del huso mitótico están asociadas con el alargamiento y acortamiento de los kMTs. Una de las características más interesantes de los cinetocoros es su capacidad de modificar el estado de sus kMTs asociados (alrededor de 20) de un estado de despolimerización de sus extremos (+) a un estado de polimerización. Esto permite a los cinetocoros de las células en prometafase mostrar "inestabilidad direccional",[69] variando entre fases persistentes de movimientos hacia el polo (poleward) o inversos (anti-poleward) que están acoplados con estados alternados de despolimerización y polimerización de los kMTs, respectivamente. Esta bi-estabilidad de los cinetocoros parece ser parte de un mecanismo para alinear los cromosomas en el ecuador del huso sin perder la conexión mecánica entre los cinetocoros y los polos del huso. Se cree que la bi-estabilidad de los cinetocoros se basa en la inestabilidad dinámica del extremo (+) de los kMTs y está controlado de forma parcial por la tensión existente en los cinetocoros. En células de mamífero en cultivo, una baja tensión en los cinetocoros promueve el cambio hacia la despolimerización de kMTs, y una alta tensión promueve el cambio hacia polimerización de los kMTs.[70] [71]
Las proteínas del cinetocoro y proteínas que se unen a los extremos (+) de los MTs (denominadas genéricamente +TIPs) regulan el movimiento del cinetocoro mediante la regulación de la dinámica de los extremos (+) de los kMTs.[72] Sin embargo, la interfaz cinetocoro-microtúbulo es muy dinámica, y algunos de estas proteínas parecen ser componentes bona fide de ambas estructuras. Dos clases de proteínas parecen ser particularmente importantes: kinesinas que funcionan como despolimerasas, tales como las kinesinas KinI; y proteínas que se unen a extremos (+) de MTs, +TIPs, que promueven la polimerización, quizás antagonizando el efecto de las despolimerasas.[73]
- Las kinesinas KinI se denominan así porque tienen un dominio motor interno, que utiliza ATP para promover la despolimerización del polímero de tubulina. En vertebrados, la kinesina KinI más importante que controla la dinámica del ensamblaje del extremo (+) es MCAK.[74] Sin embargo, parece que no es la única implicada.
- Hay dos clases de +TIPs con funciones en el cinetocoro.
- La primera incluye la proteína adenomatous polyposis coli (APC) y su proteína asociada EB1, que necesitan la presencia de MTs para localizarse en los cinetocoros. Ambas son necesarias para que la segregación cromosómica ocurra correctamente.[75] EB1 se une sólo a MTs que están en fase de polimerización, lo que sugiere que favorece la estabilización de los kMTs durante esta fase.
- Un segundo grupo de +TIPs incluye proteínas que pueden localizarse en los cinetocoros incluso en ausencia de MTs. En este grupo hay dos que han atraído mucho interés: CLIP-170 y sus proteínas asociadas CLASPs (CLIP-associated proteins). El papel de CLIP-170 en los cinetocoros es desconocido, pero la expresión de un mutante dominante negativo produce un retraso en prometafase,[76] lo que sugiere que tiene un papel activo en el alineamiento cromosómico. Las proteínas CLASPs son necesarias para el alineamiento cromosómico y el mantenimiento de un huso mitótico bipolar en Drosophila, humanos y levaduras.[77] [2]
Referencias
- ↑ Albertson, D.G.; Thomson, J.N. (1993), «Segregation of holocentric chromosomes at meiosis in the nematode, Caenorhabditis elegans», Chromosome Research 1 (1): 15–26, http://www.springerlink.com/index/L068213752103442.pdf
- ↑ a b c d e Maiato, H.; Deluca, J.; Salmon, E.D.; Earnshaw, W.C. (2004), «The dynamic kinetochore-microtubule interface», Journal of Cell Science 117 (23): 5461–5477, doi:, http://jcs.biologists.org/cgi/reprint/117/23/5461.pdf
- ↑ Mitchison, T.; Kirschner, M. (1984), «Dynamic instability of microtubule growth», Nature 312 (5991): 237–242, doi:, http://www.rpgroup.caltech.edu/courses/aph161/Handouts/Mitchison1984.pdf
- ↑ a b Brinkley, B.R.; Stubblefield, E. (1966), «The fine structure of the kinetochore of a mammalian cell in vitro», Chromosoma 19 (1): 28–43, doi:, http://www.springerlink.com/index/LX73M7135334577G.pdf
- ↑ Jokelainen, P.T. (1967), «The ultrastructure and spatial organization of the metaphase kinetochore in mitotic rat cells», J Ultrastruct Res 19 (1): 19–44, doi:, http://www.ncbi.nlm.nih.gov/pubmed/5339062
- ↑ Rieder, C.L. (1982), «The formation, structure, and composition of the mammalian kinetochore and kinetochore fiber», Int Rev Cytol 79: 1–58, doi:, http://www.ncbi.nlm.nih.gov/pubmed/6185450
- ↑ McEwen, B.F.; Hsieh, C.E.; Mattheyses, A.L.; Rieder, C.L. (1998), «A new look at kinetochore structure in vertebrate somatic cells using high-pressure freezing and …», Chromosoma 107 (6): 366–375, doi:, http://www.springerlink.com/index/K1NK8GY1E2DTC9MP.pdf
- ↑ Brenner, S.; Pepper, D.; Berns, M.W.; Tan, E.; Brinkley, B.R. (1981), «Kinetochore structure, duplication, and distribution in mammalian cells: analysis by human autoantibodies from scleroderma patients», The Journal of Cell Biology 91 (1): 95–102, doi:, PMID 7298727, http://www.jcb.org/cgi/reprint/91/1/95.pdf
- ↑ Pluta, A.F.; MacKay, A.M.; Ainsztein, A.M.; Goldberg, I.G.; Earnshaw, W.C. (1995), «The Centromere: Hub of Chromosomal Activities», Science 270 (5242): 1591, doi:, http://www.sciencemag.org/cgi/content/abstract/270/5242/1591
- ↑ Palmer, D.K.; O'Day, K.; Trong, H.L.; Charbonneau, H.; Margolis, R.L. (1991), «Purification of the Centromere-Specific Protein CENP-A and Demonstration that it is a Distinctive …», Proceedings of the National Academy of Sciences 88 (9): 3734–3738, doi:, http://www.pnas.org/cgi/reprint/88/9/3734.pdf
- ↑ Howman, E.V.; Fowler, K.J.; Newson, A.J.; Redward, S.; MacDonald, A.C.; Kalitsis, P.; Choo, K.H.A. (2000), «Early disruption of centromeric chromatin organization in centromere protein A (Cenpa) null mice», Proceedings of the National Academy of Sciences 97 (3): 1148–1153, doi:, http://www.pnas.org/cgi/content/full/97/3/1148
- ↑ Oegema, K.; Desai, A.; Rybina, S.; Kirkham, M.; Hyman, A.A. (2001), «Functional Analysis of Kinetochore Assembly in Caenorhabditis elegans», The Journal of Cell Biology 153 (6): 1209–1226, doi:, http://www.jcb.org/cgi/content/full/153/6/1209
- ↑ Van Hooser, A.A.; Ouspenski, I.I.; Gregson, H.C.; Starr, D.A.; Yen, T.J.; Goldberg, M.L.; Yokomori, K.; Earnshaw, W.C. et ál. (2001), «Specification of kinetochore-forming chromatin by the histone H3 variant CENP-A», Journal of Cell Science 114 (19): 3529–3542, http://jcs.biologists.org/cgi/content/full/114/19/3529
- ↑ Fukagawa, T.; Mikami, Y.; Nishihashi, A.; Regnier, V.; Haraguchi, T.; Hiraoka, Y.; Sugata, N.; Todokoro, K. et ál. (2001), «CENP-H, a constitutive centromere component, is required for centromere targeting of CENP-C in …», The EMBO Journal 20: 4603–4617, doi:, http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=125570
- ↑ Goshima, G.; Kiyomitsu, T.; Yoda, K.; Yanagida, M. (2003), «… protein hMis12, essential for equal segregation, is independent of CENP-A loading pathway G. …», The Journal of Cell Biology 160 (1): 25–39, doi:, http://www.jcb.org/cgi/content/full/160/1/25
- ↑ a b c d Wigge, Philip A.; Kilmartin, John V. (2001), «The Ndc80p Complex from Saccharomyces cerevisiae Contains Conserved Centromere Components and Has a Function in Chromosome Segregation», The Journal of Cell Biology 152 (2): 349–360, doi:, PMID 11266451, http://www.jcb.org/cgi/content/full/152/2/349
- ↑ a b c d Deluca, J.G.; Moree, B.; Hickey, J.M.; Kilmartin, J.V.; Salmon, E.D. (2002), «HNuf2 inhibition blocks stable kinetochore-microtubule attachment and induces mitotic cell death in HeLa cells», The Journal of Cell Biology 159 (4): 549–555, doi:, http://www.jcb.org/cgi/content/full/159/4/549
- ↑ a b Cheeseman, I.M.; Niessen, S.; Anderson, S.; Hyndman, F.; Yates, J.R.; Oegema, K.; Desai, A. (2004), «A conserved protein network controls assembly of the outer kinetochore and its ability to sustain …», Genes & Development 18 (18): 2255–2268, doi:, http://www.genesdev.org/cgi/content/full/18/18/2255
- ↑ Rattner, J.B.; Rao, A.; Fritzler, M.J.; Valencia, D.W.; Yen, T.J. (1993), «CENP-F is a. Ca 400 kDa kinetochore protein that exhibits a cell-cycle dependent localization», Cell Motil Cytoskeleton 26 (3): 214–26, doi:, http://www.ncbi.nlm.nih.gov/pubmed/7904902
- ↑ Liao, H. (1995), «CENP-F is a protein of the nuclear matrix that assembles onto kinetochores at late G2 and is rapidly …», The Journal of Cell Biology 130 (3): 507–518, doi:, http://www.jcb.org/cgi/reprint/130/3/507.pdf
- ↑ a b c d e «Microtubule-dependent Changes in Assembly of Microtubule Motor Proteins and Mitotic Spindle …», Molecular Biology of the Cell 12 (7): 1995–2009, 2001, http://www.molbiolcell.org/cgi/content/full/12/7/1995
- ↑ a b King, S.M. (2000), «The dynein microtubule motor», BBA-Molecular Cell Research 1496 (1): 60–75, http://linkinghub.elsevier.com/retrieve/pii/S0167488900000094
- ↑ a b Howell, B.J.; Moree, B.; Farrar, E.M.; Stewart, S.; Fang, G.; Salmon, E.D. (2004), «Spindle Checkpoint Protein Dynamics at Kinetochores in Living Cells», Current Biology 14 (11): 953–964, doi:, http://www.stanford.edu/group/fanglab/science/publications/22.pdf
- ↑ a b c Shah, J.V.; Botvinick, E.; Bonday, Z.; Furnari, F.; Berns, M.; Cleveland, D.W. (2004), «Dynamics of Centromere and Kinetochore Proteins Implications for Checkpoint Signaling and Silencing», Current Biology 14 (11): 942–952, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0960982204003811
- ↑ Tirnauer, Jennifer S.; Canman, Julie C.; Salmon, E.D.; Mitchison, Timothy J. (2002), «EB1 Targets to Kinetochores with Attached, Polymerizing Microtubules», Molecular Biology of the Cell 13 (12): 4308–4316, doi:, PMID 12475954, http://www.molbiolcell.org/cgi/content/full/13/12/4308
- ↑ Kaplan, K.B.; Burds, A.A.; Swedlow, J.R.; Bekir, S.S.; Sorger, P.K.; Näthke, I.S. (2001), «A role for the Adenomatous Polyposis Coli protein in chromosome segregation», Nature Cell Biology 3: 429–432, doi:, http://www.nature.com/ncb/journal/v3/n4/abs/ncb0401_429.html
- ↑ Joseph, J.; Liu, S.T.; Jablonski, S.A.; Yen, T.J.; Dasso, M. (2004), «The RanGAP1-RanBP2 Complex is Essential for Microtubule-Kinetochore Interactions in Vivo», Current Biology 14 (7): 611–617, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0960982204002106
- ↑ Salina, Davide; Enarson, Paul; Rattner, J.B.; Burke, Brian (2003), «Nup358 integrates nuclear envelope breakdown with kinetochore assembly», The Journal of Cell Biology 162 (6): 991–1002, doi:, PMID 12963708
- ↑ Ohta S, Bukowski-Wills JC, Sanchez-Pulido L, Alves Fde L, Wood L, Chen ZA, Platani M, Fischer L, Hudson DF, Ponting CP, Fukagawa T, Earnshaw WC, Rappsilber J (September 2010), «The protein composition of mitotic chromosomes determined using multiclassifier combinatorial proteomics», Cell 142 (5): 810–21, doi:, PMID 20813266
- ↑ McEwen, B.F.; Heagle, A.B.; Cassels, G.O.; Buttle, K.F.; Rieder, C.L. (1997), «Kinetochore Fiber Maturation in PtK1 Cells and Its Implications for the Mechanisms of Chromosome …», The Journal of Cell Biology 137 (7): 1567–1580, doi:, http://www.jcb.org/cgi/content/full/137/7/1567
- ↑ a b Nicklas, R.B.; Kubai, D.F. (1985), «Microtubules, chromosome movement, and reorientation after chromosomes are detached from the spindle …», Chromosoma 92 (4): 313–324, doi:, http://www.springerlink.com/index/U2714G8M11405507.pdf
- ↑ Mayor, T.; Meraldi, P.; Stierhof, Y.D.; Nigg, E.A.; Fry, A.M. (1999), «Protein kinases in control of the centrosome cycle», FEBS Letters 452 (1-2): 92–95, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0014579399005347
- ↑ a b Kirschner, M.; Mitchison, T. (1986), «Beyond self-assembly: from microtubules to morphogenesis», Cell 45 (3): 329–342, doi:, http://mitchison.med.harvard.edu/publications/CellV45p329.pdf
- ↑ Holy, T. E.; Leibler, S. (1994), «Dynamic instability of microtubules as an efficient way to search in space», Proceedings of the National Academy of Sciences of the United States of America 91 (12): 5682–5685, doi:, PMID 8202548, http://www.pnas.org/cgi/reprint/91/12/5682.pdf
- ↑ Hayden, J.H. (1990), «Kinetochores capture astral microtubules during chromosome attachment to the mitotic spindle: direct …», The Journal of Cell Biology 111 (3): 1039–1045, doi:, http://www.jcb.org/cgi/reprint/111/3/1039.pdf
- ↑ Nicklas, R.B. (1997), «How Cells Get the Right Chromosomes», Science 275 (5300): 632, doi:, http://www.blc.arizona.edu/courses/mcb572/PDFs-2005/0307Niklas.pdf
- ↑ Loncarek, J.; Kisurina-evgenieva, O.; Vinogradova, T.; Hergert, P.; La Terra, S.; Kapoor, T.M.; Khodjakov, A. (2007), «The centromere geometry essential for keeping mitosis error free is controlled by spindle forces», Nature 450 (7170): 745, doi:, http://www.nature.com/nature/journal/v450/n7170/abs/nature06344.html
- ↑ Dewar, H.; Tanaka, K.; Nasmyth, K.; Tanaka, T.U. (2004), Tension between two kinetochores suffices for their bi-orientation on the mitotic spindle, http://www.nature.com/nature/journal/v428/n6978/abs/nature02328_ja.html?lang=en
- ↑ Echeverri, C.J.; Paschal, B.M.; Vaughan, K.T.; Vallee, R.B. (1996), «Molecular characterization of the 50-kD subunit of dynactin reveals function for the complex in chromosome alignment and spindle organization during mitosis», The Journal of Cell Biology 132 (4): 617–633, doi:, PMID 8647893, http://www.jcb.org/cgi/reprint/132/4/617.pdf
- ↑ Sharp, D.J.; Rogers, G.C.; Scholey, J.M. (2000), «Cytoplasmic dynein is required for poleward chromosome movement during mitosis in Drosophila embryos», Nature Cell Biology 2: 922–930, doi:, http://www.mcb.ucdavis.edu/faculty-labs/scholey/Journal%20Papers/sharp%20nature%202000.pdf
- ↑ Banks, J.D.; Heald, R. (2001), «Chromosome movement: Dynein-out at the kinetochore», Current Biology 11 (4): 128–131, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0960982201000598
- ↑ Howell, B.J.; McEwen, B.F.; Canman, J.C.; Hoffman, D.B.; Farrar, E.M.; Rieder, C.L.; Salmon, E.D. (2001), «Cytoplasmic dynein/dynactin drives kinetochore protein transport to the spindle poles and has a role in mitotic spindle checkpoint inactivation», The Journal of Cell Biology 155 (7): 1159–1172, doi:, PMID 11756470
- ↑ Cooke, C.A.; Schaar, B.; Yen, T.J.; Earnshaw, W.C. (1997), «Localization of CENP-E in the fibrous corona and outer plate of mammalian kinetochores from …», Chromosoma 106 (7): 446–455, doi:, http://www.springerlink.com/index/87BBGWBXFWYUHWHP.pdf
- ↑ Weaver, Beth A.A.; Bonday, Zahid Q.; Putkey, Frances R.; Kops, Geert J.P.L.; Silk, Alain D.; Cleveland, Don W. (2003), «Centromere-associated protein-E is essential for the mammalian mitotic checkpoint to prevent aneuploidy due to single chromosome loss», The Journal of Cell Biology 162 (4): 551–563, doi:, PMID 12925705, http://www.uvm.edu/~biology/Classes/263/jcb2003-5.pdf
- ↑ Mitchison, T.J. (1988), «Microtubule Dynamics and Kinetochore Function in Mitosis», Annual Reviews in Cell Biology 4 (1): 527–545, doi:
- ↑ a b c He, X.; Rines, D.R.; Espelin, C.W.; Sorger, P.K. (2001), «Molecular Analysis of Kinetochore-Microtubule Attachment in Budding Yeast», Cell 106 (2): 195–206, doi:, http://www.cell.com/cgi/content/full/106/2/195/
- ↑ a b Westermann, Stefan; Cheeseman, Iain M.; Anderson, Scott; Yates, John R.; I. I. I.; Drubin, David G.; Barnes, Georjana (2003), «Architecture of the budding yeast kinetochore reveals a conserved molecular core», The Journal of Cell Biology 163 (2): 215, doi:, PMID 14581449, http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2173538
- ↑ a b De Wulf, P.; McAinsh, A.D.; Sorger, P.K. (2003), «Hierarchical assembly of the budding yeast kinetochore from multiple subcomplexes», Genes & Development 17 (23): 2902–2921, doi:, http://www.genesdev.org/cgi/content/full/17/23/2902
- ↑ Goh, P.Y.; Kilmartin, J.V. (1993), «NDC10: a gene involved in chromosome segregation in Saccharomyces cerevisiae», The Journal of Cell Biology 121 (3): 503–12, doi:, PMID 8486732, http://www.ncbi.nlm.nih.gov/pubmed/8486732
- ↑ Nabetani, A.; Koujin, T.; Tsutsumi, C.; Haraguchi, T.; Hiraoka, Y. (2001), «A conserved protein, Nuf2, is implicated in connecting the centromere to the spindle during …», Chromosoma 110 (5): 322–334, doi:, http://www.springerlink.com/index/3N1UGJMQVL60L120.pdf
- ↑ a b Howe, Mary; McDonald, Kent L.; Albertson, Donna G.; Meyer, Barbara J. (2001), «HIM-10 is Required for Kinetochore Structure and Function on Caenorhabditis elegans Holocentric Chromosomes», The Journal of Cell Biology 153 (6): 1227–1238, doi:, PMID 11402066, http://www.jcb.org/cgi/content/full/153/6/1227
- ↑ a b c Martin-lluesma, Silvia; Stucke, Volker M.; Nigg, Erich A. (2002), «Role of Hec1 in Spindle Checkpoint Signaling and Kinetochore Recruitment of Mad1/Mad2», Science 297 (5590): 2267–2270, doi:, PMID 12351790, http://www.sciencemag.org/cgi/content/abstract/297/5590/2267
- ↑ a b c McCleland, M.L.; Gardner, R.D.; Kallio, M.J.; Daum, J.R.; Gorbsky, G.J.; Burke, D.J.; Stukenberg, P.T. (2003), «The highly conserved Ndc80 complex is required for kinetochore assembly, chromosome congression, and …», Genes & Development 17 (1): 101–114, doi:, http://www.genesdev.org/cgi/content/full/17/1/101
- ↑ Zheng, L.; Chen, Y.; Lee, W.H. (1999), «Hec1p, an Evolutionarily Conserved Coiled-Coil Protein, Modulates Chromosome Segregation through …», Molecular and Cellular Biology 19 (8): 5417–5428, http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=84384
- ↑ Wei, Ronnie R.; Al-bassam, Jawdat; Harrison, Stephen C. (2007), «The Ndc80/HEC1 complex is a contact point for kinetochore-microtubule attachment», Nature Structural & Molecular Biology 14 (1): 54–59, doi:, https://crystal.harvard.edu/PDFs/wei_et_al_nsmb_2007.pdf
- ↑ Courtwright, A.M.; He, X. (2002), «Dam1 is the Right One Phosphoregulation of Kinetochore Biorientation», Developmental Cell 3 (5): 610–611, doi:, http://linkinghub.elsevier.com/retrieve/pii/S1534580702003325
- ↑ a b Cimini, D.; Moree, B.; Canman, J.C.; Salmon, E.D. (2003), «� Merotelic kinetochore orientation occurs frequently during early mitosis in mammalian tissue cells …», Journal of Cell Science 116 (20): 4213–4225, doi:, http://jcs.biologists.org/cgi/reprint/jcs.00716v1.pdf
- ↑ Adams, R.R.; Carmena, M.; Earnshaw, W.C. (2001), «Chromosomal passengers and the (aurora) ABCs of mitosis», Trends in Cell Biology 11 (2): 49–54, doi:, http://web.bio.ed.ac.uk/research/groups/earnshaw/pdf/Adams%20TiCB%2011,%2049-54.PDF
- ↑ Cheeseman, I.M.; Anderson, S.; Jwa, M.; Green, E.M.; Kang, J.; Yates, J.R.; Chan, C.S.M.; Drubin, D.G. et ál. (2002), «Phospho-Regulation of Kinetochore-Microtubule Attachments by the Aurora Kinase Ipl1p», Cell 111 (2): 163–172, doi:, http://www.cell.com/cgi/content/reprint/111/2/163
- ↑ Gautschi, Oliver; Heighway, Jim; Mack, Philip C.; Purnell, Phillip R.; Lara, Primo N.; Jr, .; Gandara, David R. (2008), «Aurora Kinases as Anticancer Drug Targets», Clinical Cancer Research 14 (6): 1639, doi:, PMID 18347165, http://clincancerres.aacrjournals.org/cgi/content/abstract/14/6/1639
- ↑ Meraldi, P.; Draviam, V.M.; Sorger, P.K. (2004), «Timing and Checkpoints in the Regulation of Mitotic Progression», Developmental Cell 7 (1): 45–60, doi:, http://microarray.princeton.edu/mb523/pdf/meraldi04.pdf
- ↑ Tang, T.T.L.; Bickel, S.E.; Young, L.M.; Orr-weaver, T.L. (1998), «Maintenance of sister-chromatid cohesion at the centromere by the Drosophila MEI-S332 protein», Genes & Development 12 (24): 3843–3856, doi:, http://www.genesdev.org/cgi/content/abstract/12/24/3843
- ↑ McGuinness, B.E.; Hirota, T.; Kudo, N.R.; Peters, J.M.; Nasmyth, K. (2005), «Shugoshin Prevents Dissociation of Cohesin from Centromeres During Mitosis in Vertebrate Cells», PLoS Biol 3 (3): e86, doi:, http://biology.plosjournals.org/plosonline/?request=get-document
- ↑ Joseph, Jomon; Tan, Shyh-Han; Karpova, Tatiana S.; McNally, James G.; Dasso, Mary (2002), «SUMO-1 targets RanGAP1 to kinetochores and mitotic spindles», The Journal of Cell Biology 156 (4): 595–602, doi:, PMID 11854305, http://www.jcb.org/cgi/content/full/156/4/595
- ↑ Arnaoutov, A.; Dasso, M. (2003), «The Ran GTPase Regulates Kinetochore Function», Developmental Cell 5 (1): 99–111, doi:, http://linkinghub.elsevier.com/retrieve/pii/S1534580703001941
- ↑ Prasanth, S.G.; Prasanth, K.V.; Siddiqui, K.; Spector, D.L.; Stillman, B. (2004), «Human Orc2 localizes to centrosomes, centromeres and heterochromatin during chromosome inheritance», The EMBO Journal 23: 2651–2663, doi:, http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=449767
- ↑ Shimada, K.; Gasser, S.M. (2007), «The Origin Recognition Complex Functions in Sister-Chromatid Cohesion in Saccharomyces cerevisiae», Cell 128 (1): 85–99, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0092867406016096
- ↑ Kato, H.; Matsunaga, F.; Miyazaki, S.; Yin, L.; D'urso, G.; Tanaka, K.; Murakami, Y. (2008), «Schizosaccharomyces pombe Orc5 plays multiple roles in the maintenance of genome stability …», Cell cycle (Georgetown, Tex.) 7 (8), http://www.ncbi.nlm.nih.gov/pubmed/18414064
- ↑ Skibbens, R.V.; Skeen, V.P.; Salmon, E.D. (1993), «Directional instability of kinetochore motility during chromosome congression and segregation in mitotic newt lung cells: a push-pull mechanism», The Journal of Cell Biology 122 (4): 859–875, doi:, PMID 8349735, http://www.jcb.org/cgi/reprint/122/4/859.pdf
- ↑ Rieder, C.L.; Salmon, E.D. (1994), «Motile kinetochores and polar ejection forces dictate chromosome position on the vertebrate mitotic spindle», The Journal of Cell Biology 124 (3): 223, doi:, PMID 8294508, http://www.jcb.org/cgi/reprint/124/3/223.pdf
- ↑ Skibbens, R.V. (1995), «Kinetochore motility after severing between sister centromeres using laser microsurgery: evidence that kinetochore directional instability and position is regulated by tension», Journal of Cell Science 108 (7): 2537–2548, http://jcs.biologists.org/cgi/reprint/108/7/2537.pdf
- ↑ Askham, J. M.; Vaughan, K. T.; Goodson, H. V.; Morrison, E. E. (2002), «Evidence That an Interaction between EB1 and p150Glued is Required for the Formation and Maintenance of a Radial Microtubule Array Anchored at the Centrosome», Molecular Biology of the Cell 13 (10): 3627–3645, doi:, PMID 12388762, http://www.molbiolcell.org/cgi/content/full/13/10/3627
- ↑ Schuyler, S.C.; Pellman, D. (2001), «Microtubule “Plus-End-Tracking Proteins” the End is Just the Beginning», Cell 105 (4): 421–424, doi:, http://www.cell.com/cgi/content/full/105/4/421/
- ↑ Howard, J.; Hyman, A.A. (2003), «Dynamics and mechanics of the microtubule plus end: cytoskeleton», Nature(London) 422 (6933): 753–758, http://cat.inist.fr/?aModele=afficheN
- ↑ Green, R.A.; Wollman, R.; Kaplan, K.B. (2005), «APC and EB1 Function Together in Mitosis to Regulate Spindle Dynamics and Chromosome Alignment», Molecular Biology of the Cell 16 (10): 4609–4622, doi:, http://www.molbiolcell.org/cgi/content/full/16/10/4609
- ↑ Dujardin, D.; Wacker, U.I.; Moreau, A.; Schroer, T.A.; Rickard, J.E.; De Mey, J.R. (1998), «Evidence for a Role of CLIP-170 in the Establishment of Metaphase Chromosome Alignment», The Journal of Cell Biology 141 (4): 849–862, http://www.jcb.org/cgi/content/full/141/4/849
- ↑ Maiato, H.; Fairley, E.A.L.; Rieder, C.L.; Swedlow, J.R.; Sunkel, C.E.; Earnshaw, W.C. (2003), «Human CLASP1 is an Outer Kinetochore Component that Regulates Spindle Microtubule Dynamics», Cell 113 (7): 891–904, doi:, http://linkinghub.elsevier.com/retrieve/pii/S0092867403004653
Véase también
- Huso mitótico
- Punto de control
- Checkpoint de mitosis
- Cromosoma
- Ciclo celular
- Mitosis
- Microtúbulo
Enlaces externos
Categorías:- Citogenética
- Cromosomas
Wikimedia foundation. 2010.