- Poro nuclear
-
Poro nuclear
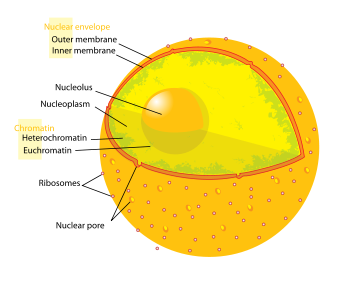
Los poros nucleares son grandes complejos de proteínas que atraviesan la envoltura nuclear, la cual es una doble membrana que rodea al núcleo celular, presente en la mayoría de los eucariontes. Hay cerca de 2000 complejos de poro en la envoltura nuclear en la célula de un vertebrado, pero varía dependiendo del número de transcripciones de la célula. Las proteínas que forman los complejos de poro nucleares son conocidas como nucleoporinas. Cerca de la mitad de las nucleoporinas contienen comúnmente una estructura terciaria alfa solenoide o beta hélice, o en algunos casos ambas como dominios proteicos separados. La otra mitad muestra características estructurales típicas de proteínas "nativamente no dobladas", por ejemplo son proteínas altamente flexibles que carecen de estructura secundaria ordenada[1] . Estas proteínas desordenadas son las nucleoporinas FG, llamadas así por su secuencia aminoacídica que contiene varias repeticiones del péptido fenilalanina-glicina[2] .
Los poros nucleares permiten el transporte de moléculas solubles en agua a través de la envoltura nuclear. Este transporte incluye el movimiento de ARN y ribosomas desde el núcleo al citoplasma, y movimiento de proteínas (tales como ADN polimerasa y lamininas), carbohidratos, moléculas de señal y lípidos hacia el núcleo. Es notable que el complejo de poro nuclear (CPN o NPC en inglés) puede conducir activamente 1000 translocaciones por complejo por segundo. Aunque las moléculas pequeñas pasan por difusión simple a través de los poros, las moléculas de mayor tamaño pueden ser reconocidas mediante secuencias de señal específicas y luego difundidas con la ayuda de las nucleoporinas hacia o desde el núcleo. Esto es conocido como el ciclo RAN. Cada una de las ocho subunidades proteicas que rodean el poro verdadero (el anillo externo) proyecta una proteína con forma de radio hacía el canal del poro. El centro del poro muchas veces parece que tuviera una estructura parecida a un tapón. Aún no se sabe sí esto corresponde a un tapón verdadero o es simplemente carga atrapada durante el transito.
Contenido
Tamaño y complejidad
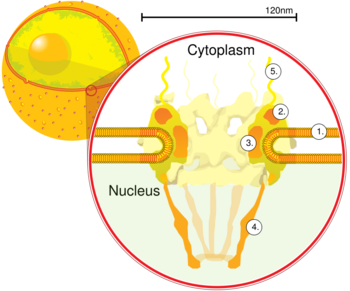
El complejo de poro completo tiene un diámetro de cerca de 120 nm, el diámetro de la abertura es cercano a los 50 nm de ancho, y su profundidad es de cerca de 200 nm. La masa molecular del complejo de poro es cerca de 125 millones de daltons y contiene aproximadamente 50 componentes proteicos diferentes.
El dominio proteico de nucleoproteínas FG que rodea al poro nuclear, responde a interacciones biofísicas con el fin de proveer transporte entre el núcleo y el citoplasma. En condiciones normales, la sección que contiene las nucleoporinas FG, se colapsa reversiblemente dependiendo de reguladores del transporte nuclear, como el Ran guanosina de trifosfato.[3] Estas nucleoporinas están por lo general, localizadas en el lado de la periferia citoplasmática y consisten en motivos repetitivos de Phe-Gly.
Transporte a través del complejo de poro nuclear
Las partículas pequeñas (< 50 kDa) son capaces de pasar a través del complejo de poro nuclear mediante difusión pasiva. También pueden pasar partículas más grandes a través del diámetro grande del poro, pero a tasas casi insignificativas[4] El paso eficiente a través del complejo requiere varios factores proteicos.[5] . La sencillez del transporte por los poros nucleares es facilitada por receptores en los dominios FG llamados Carioferinas, los cuales son requeridos para el transporte núcleo-citoplásmico de moléculas mayores a 40 kDa. En la ausencia de estos receptores, abreviados Kaps, los dominios FG imponen una barrera física que impide el paso de macromoléculas a través del poro nuclear.[3] Las carioferinas, las cuales pueden actuar como importinas o exportinas, son parte de la super familia de la importina -β las que comparten en su totalidad una estructura tridimensional similar.
Han sido sugeridos tres modelos para explicar los mecanismos de translocación:
- Gradientes de afinidad a través del tapón (plug) central.
- Afinidad browniana de apertura.
- Fase selectiva.
Importación de proteínas solubles
Cualquier partícula que porte una señal de localización nuclear (NLS) será dirigida por el rápido y eficiente transporte a través del poro. Muchas señales de localización nuclear son conocidas, generalmente contienen una secuencia de aminoácidos conservada con residuos básicos tales como PKKKRKV. Cualquier material con una señal de localización nuclear será llevada por las importinas hacia el núcleo.
El esquema clásico para la importación de partículas con señal de localización nuclear comienza primero con la α-importina uniéndose a la secuencia de la señal de localización nuclear, y actúa como un puente para unir la β-importina. Luego el complejo carga-βimportina—αimportina es dirigida hacia el poro nuclear y difunde a través de él. Una vez que el complejo está en el núcleo, se une RanGTP a la βimportina y la desplaza del complejo. Luego la proteína de suceptibilidad de apoptosis celular (CAS), una exportina que está unida a RanGTP en el núcleo, separa la α-importina de la carga. La proteína de señal de localización nuclear se encuentra de esta manera libre en el nucleoplasma. Los complejos βimportina-RanGTP e αimportina-CAS-RanGTP difunden de vuelta hacia el citoplasma donde los GTPs son hidrolizados a GDP llevando a la liberación de βimportina y αimportin que vuelve a estar disponible para una nueva importación de proteínas de señal de localización nuclear.
Aunque la carga pase a través del poro con la asistencia de proteínas chaperonas, la translocación a través del poro no es por sí misma dependiente de energía. Sin embargo, el ciclo completo de importación necesita la hidrólisis de 2 GTPs y por lo tanto es energía dependiente y tiene que ser considerada como transporte activo. El ciclo de importación funciona gracias al gradiente de RanGTP núcleo-citoplasmático. Este gradiente surge de la localización nuclear exclusiva de RanGEFs, proteínas que cambian GDP a GTP en moléculas Ran. Por lo tanto hay una concentración elevada de RanGTP en el núcleo comparada con el citoplasma.
Exportación de proteínas
Algunas moléculas nucleares necesitan ser exportadas desde el núcleo al citoplasma, como las subunidades ribosomales y ARNs mensajeros. Por lo tanto hay un mecanismo de exportación similar al de importación.
En el esquema clásico de exportación, las proteínas con una secuencia de exportación nuclear (NES) se pueden unir en el núcleo para formar un complejo heterotrimérico con una exportina y RanGTP (por ejemplo la exportina CRM1!. El complejo puede difundir al citoplasma donde el GTP es hidrolizado y la proteína de secuencia de exportación nuclear es liberada. El complejo CRM1-RanGDP difunde de vuelta hacia el núcleo donde el GDP es cambiado a GTP por las RanGEFs. Este porceso también es dependiente de energía ya que consume GTP. La exportación con CRM1 puede ser inhibida por la leptomicina B.
Referencias
- ↑ Denning D, Patel S, Uversky V, Fink A, Rexach M (2003). «Disorder in the nuclear pore complex: the FG repeat regions of nucleoporins are natively unfolded» Proc Natl Acad Sci U S A. Vol. 100. n.º 5. pp. 2450-5. PMID 12604785.
- ↑ Peters R (2006). «Introduction to nucleocytoplasmic transport: molecules and mechanisms» Methods Mol Biol. Vol. 322. pp. 235-58. PMID 16739728.
- ↑ a b Lim RY, Fahrenkrog B, et al. Nanomechanical Basis of Selective Gating by the Nuclear Pore Complex. Science. 2007 Oct 26. 318(5850):640-643. Disponible en la World Wide Web: PMID: 17916694 Último acceso 22 Noviembre 2007
- ↑ Rodriguez M, Dargemont C, Stutz F (2004). «Nuclear export of RNA» Biol Cell. Vol. 96. n.º 8. pp. 639-55. PMID 15519698.
- ↑ Reed R, Hurt E (2002). «A conserved mRNA export machinery coupled to pre-mRNA splicing» Cell. Vol. 108. n.º 4. pp. 523-31. PMID 11909523.
Categoría: Núcleo celular
Wikimedia foundation. 2010.