- Repetición rica en leucina
-
Repetición rica en leucina
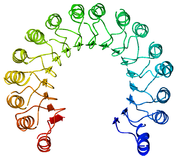
Una repetición rica en leucina (LRR por sus siglas en inglés) es un motivo estructural de proteínas que forma un plegamiento en herradura α/β.[1] [2] Se compone de la repetición de 20-30 aminoácidos que se tratan generalmente de residuos del aminoácido hidrofóbico leucina.
Normalmente, cada unidad repetida tiene una estructura tipo lámina beta-giro-hélice alfa, y el dominio ensamblado, compuesto por muchas de estas repeticiones, tiene la forma de una herradura con un interior de láminas paralelas y un exterior de arreglos de hélices alfa. Una cara de la lámina beta y un lado del arreglo de hélices están expuestos al solvente y por ende poseen residuos hidrofílicos. La región entre las hélices y las láminas beta forma el núcleo hidrofóbico de la proteína y está estrechamente empaquetado con residuos de leucina.
Las repeticiones ricas en leucina están con frecuencia involucrados en la formación de las interacciones proteína-proteína.[3] [4]
Contenido
Ejemplos
Los motivos de repeticiones ricas en leucina se han identificado en un gran número de proteínas no relacionadas funcionalmente.[5] El ejemplo más conocido es el inhibidor de la ribonucleasa, pero otras proteínas tales como el regulador de tropomiosina, tropomodulina y los receptores tipo Toll también comparten este motivo. De hecho, el receptor tipo Toll posee diez motivos de repeticiones ricas en leucina sucesivos que sirven para unirse al antígeno.
Aunque la proteína con repeticiones ricas en leucina canónica contiene aproximadamente una hélice para cada lámina beta, las variantes que forman los plegamientos de superhélice beta-alfa a veces en lugar de hélices poseen largos bucles que unen sucesivas láminas beta.
Referencias
- ↑ Kobe B, Deisenhofer J (October de 1994). «The leucine-rich repeat: a versatile binding motif» Trends Biochem. Sci.. Vol. 19. n.º 10. pp. 415–21. PMID 7817399.
- ↑ Enkhbayar P, Kamiya M, Osaki M, Matsumoto T, Matsushima N (February de 2004). «Structural principles of leucine-rich repeat (LRR) proteins» Proteins. Vol. 54. n.º 3. pp. 394–403. DOI 10.1002/prot.10605. PMID 14747988.
- ↑ Kobe B, Kajava AV (diciembre de 2001). «The leucine-rich repeat as a protein recognition motif» Curr. Opin. Struct. Biol.. Vol. 11. n.º 6. pp. 725–32. PMID 11751054.
- ↑ Gay NJ, Packman LC, Weldon MA, Barna JC (octubre de 1991). «A leucine-rich repeat peptide derived from the Drosophila Toll receptor forms extended filaments with a beta-sheet structure» FEBS Lett.. Vol. 291. n.º 1. pp. 87–91. PMID 1657640.
- ↑ Rothberg JM, Jacobs JR, Goodman CS, Artavanis-Tsakonas S (diciembre de 1990). «slit: an extracellular protein necessary for development of midline glia and commissural axon pathways contains both EGF and LRR domains» Genes Dev.. Vol. 4. n.º 12A. pp. 2169–87. PMID 2176636.
Lecturas complementarias
- R. K. Wierenga (2001). «The TIM-barrel fold: a versatile framework for efficient enzymes» FEBS Lett.. Vol. 492. n.º 3. pp. 192–198. DOI 10.1016/S0014-5793(01)02236-0.
Enlaces externos
- Plegamiento LRR en la base de datos SCOP
- Arquitetura en herradura alfa/beta en CATH
- LRRML: base de datos conformacional de repeticiones ricas en leucina
Categoría: Estructura de las proteínas
Wikimedia foundation. 2010.