- Bloqueo de genes
-
El bloqueo de genes (en inglés, gene knockout) es una técnica genética que consiste en suprimir la expresión de un gen específico en un organismo (un ratón, una planta, una levadura...), sustituyendo el gen original en su locus por una versión modificada del mismo, a la que se ha extraído uno o varios exones para generar una versión no funcional, incapaz de producir la proteína que codificaba el gen original.[1] [2] De esta forma, se obtienen organismos que no expresan el gen diana en un tejido específico o en el organismo completo, y que se denominan organismos knockout, como es el caso de los ratones knockout.
Existen diferentes técnicas de biología molecular para interferir con la expresión de un gen. Estas técnicas varían en las herramientas utilizadas, en el tiempo necesario para conseguir un resultado positivo y en la especificidad del mismo. Dentro de estas técnicas se encuentran:
- el knockout (en español, noqueo) de genes es una técnica en la que el gen endógeno se extrae: se intercambia por recombinación homóloga por una versión mutada (que se introduce). Esta versión mutada puede eliminar prácticamente toda la secuencia del gen o bien dejar la expresión de los primeros exones, pero es preferible no dejar casi nada; esta técnica es la que se describe en el apartado "Metododología". Suelen requerirse varios meses para obtener individuos modificados (sobre todo en el caso de los ratones), aunque presentan la ventaja de ser muy específicos: estos organismos únicamente presentan carencia del gen diana.
- en la transgénesis, se introducen secuencias dentro del gen original que lo alteran, para producir modificaciones que generen proteínas no funcionales. En este caso, los organismos obtenidos se denominan, por ejemplo, "ratones transgénicos". Suele ser una técnica más rápida, cuyo inconveniente es que el gen endógeno aún está presente, y puede producir una proteína, aunque mutada, que puede interferir con otras moléculas de la vía a la que pertenece. Además, puede producirse por azar una nueva mutación que revierta la modificación introducida, generando de nuevo la proteína original, suceso que tiene lugar con una cierta frecuencia.
- se pueden obtener mutantes utilizando un producto mutagénico (como el EMS, por etil metanosulfanato), que producen mutaciones al azar, y seleccionar luego organismos que lleven codones "STOP" en la secuencia de interés. Es un método rápido de obtener mutantes, aunque no es específico para la secuencia diana.
- se pueden introducir en el genoma de un organismo secuencias de ADN que expresen moléculas de ARN interferente. Estas moléculas interfieren de forma específica con la expresión del gen diana, produciendo generalmente una disminución de la expresión (que se denomina "knockdown" por este motivo). Esta técnica es más rápida que la obtención de un organismo "knockout", pero no produce una eliminación completa de la expresión y además la secuencia introducida que codifica para el ARN interferente puede también sufrir mutaciones, inactivando el efecto deseado.
Por estas razones, los organismos knockouts son los modelos preferidos a la hora de estudiar la función de un gen.
Metodología de la técnica knockout
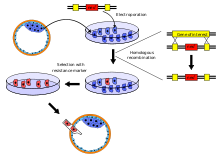
- El gen modificado (que carece la mayor parte de la secuencia del gen original) se introduce en un plásmido, para generar un vector direccionador (targeting vector). En dicho vector, el gen modificado está controlado por un promotor que permite la expresión del gen de forma bien constitutiva (continua durante todo el desarrollo del animal), bien condicional (dependiendo de la presencia o no de una sustancia concreta que actúa como activador o inhibidor, como la tetraciclina; ello permite expresar el gen únicamente en una etapa del desarrollo o en un tejido concreto). Además, en el vector direccionador el gen modificado está flanqueado por fragmentos de los brazos cromosómicos colindantes del locus del gen de interés (en posición 5' y en posición 3'). Tanto el gen de interés como los brazos cromosómicos se obtienen a partir de un fragmento del genoma de la zona correspondiente, por ejemplo a partir de un BAC; el gen de interés se modifica mediante técnicas de biología molecular, y los tres componentes (el gen modificado y los brazos) se introducen (se subclonan) en el plásmido. Asimismo, el plásmido debe incluir la presencia de un marcador (normalmente la resistencia a un antibiótico) que permita seleccionar las células que han incorporado el gen modificado. Hay diferentes variantes de plásmidos disponibles, en función de la técnica concreta que se vaya a utilizar para crear el vector direccionador.
 Esquema de cruces para obtener ratones con genes bloqueados. Blastocitos que contienen células tanto no modificadas como bloqueadas, se inyectan en el útero de una madre portadora. Ésta produce una descendencia que es bien tipo salvaje (no modificados, del mismo color que el blastocito donante (gris)) o bien es una quimera (mezcla) (parcialmente bloqueada). Los ratones quimera se cruzan con un ratón normal gris. Este cruce produce una descendencia de ratones blancos (heterocigotos para el gen bloqueado) o bien grises (normales). Los ratones blancos heterocigotos se cruzan luego entre sí para obtener ratones que son homocigotos para el gen bloqueado o mas bien, suprimido.
Esquema de cruces para obtener ratones con genes bloqueados. Blastocitos que contienen células tanto no modificadas como bloqueadas, se inyectan en el útero de una madre portadora. Ésta produce una descendencia que es bien tipo salvaje (no modificados, del mismo color que el blastocito donante (gris)) o bien es una quimera (mezcla) (parcialmente bloqueada). Los ratones quimera se cruzan con un ratón normal gris. Este cruce produce una descendencia de ratones blancos (heterocigotos para el gen bloqueado) o bien grises (normales). Los ratones blancos heterocigotos se cruzan luego entre sí para obtener ratones que son homocigotos para el gen bloqueado o mas bien, suprimido.
- Una vez construido el vector direccionador (un proceso complejo que puede implicar varios meses de trabajo), éste se añade a un cultivo de células embrionarias ES (stem cells, células madre), y se incorpora a las células mediante transfección. El vector direccionador puede reconocer (por apareamiento de bases) la zona del locus de interés, de manera que mediante el mecanismo de recombinación homóloga, se puede producir un intercambio entre el vector direccionador (que recibe el gen endógeno) y el locus genómico (que recibe el gen modificado no funcional). Por este motivo, cuanto más largas sean las secuencias de los brazos de homología que flanquean al gen modificado, mayor será la probabilidad de que se produzca recombinación homóloga (y por tanto aparezcan células con el gen modificado), aunque eso aumenta la dificultad de construcción del vector direccionador.[3]
- Las células ES transfectadas se someten entonces a selección, utilizando el antibiótico correspondiente, de manera que sólo sobreviven las que hayan incorporado en su genoma el gen modificado (si el vector no se ha recombinado, se diluye tras un cierto número de divisiones celulares). Las células que sobreviven al proceso de selección se verifican mediante Southern Blot, una técnica de biología molecular que permite verificar la presencia de una molécula de ADN concreta en una muestra de ADN (el Southern blot combina la separación de fragmentos de ADN mediante electroforesis en geles de agarosa con métodos para transferir los fragmentos de ADN separados por tamaño a una membrana, que se hibrida con una sonda específica para detectar la presencia de la molécula de interés).
- Una vez verificado que las células embrionarias ES efectivamente han sustituido el gen endógeno (que ha sido extraído, de ahí el término knockout del ingles, entendiendo que uno ha llegado a derribar o interceptar al otro) por el gen modificado no funcional, algunas de estas células ES se inoculan a blastocitos, que son transplantados a hembras pseudopreñadas (en el caso de ratones). Los organismos con genes bloqueados (organismos knockout) se utilizan para estudiar el rol funcional de un determinado gen por defecto, estudiando las diferencias entre el organismo knockout e individuos normales de la misma cepa.
Un organismo knockin (con genes bloqueadores) es igual al anterior pero en lugar de eliminar un gen determinado este es reemplazado por una versión modificada del mismo, que produce una variación en su función.
En 2007, Mario Capecchi, Martin Evans, y Oliver Smithies obtuvieron el Premio Nobel en Fisiología o Medicina por el desarrollo de técnicas de modificación genética específicas y tecnología de células madre embriónicas de ratón (células ES) que, combinadas, permitieron la generación de los ratones knockout (ratones con genes bloqueados). El análisis de estos animales mutantes ha revolucionado la elucidación de la función de los genes, y estos ratones han demostrado ser un valioso modelo de numerosas enfermedades humanas.[4] [5] [6] [7] [8] [9] [10] [11] [12]
Referencias
- ↑ Capecchi MR. Altering the genome by homologous recombination. Science 1989 Jun 16;244(4910):1288-92 [1]
- ↑ Capecchi MR. The new mouse genetics: altering the genome by gene targeting. Trends Genet. 1989 5: 70–76 [2]
- ↑ Glaser S, Anastassiadis K, Stewart AF. Current issues in mouse genome engineering. Nat Genet. 2005 Nov;37(11):1187-93. [3]
- ↑ Houdebine L (2007). «Transgenic animal models in biomedical research». Methods Mol Biol 360: pp. 163–202. PMID 17172731. [4]
- ↑ Green JE, Hudson T. The promise of genetically engineered mice for cancer prevention studies. Nat Rev Cancer 2005 Mar;5(3):184-98 [5]
- ↑ McCormack E, Bruserud O, Gjertsen BT. Review: genetic models of acute myeloid leukaemia. Oncogene 2008 Jun 19;27(27):3765-79 [6]
- ↑ Øvlisen K, Kristensen AT, Tranholm M. In vivo models of haemophilia - status on current knowledge of clinical phenotypes and therapeutic interventions. Haemophilia 2008 Mar;14(2):248-59 [7]
- ↑ Boehncke WH, Schön MP. Animal models of psoriasis. Clin Dermatol. 2007 Nov-Dec;25(6):596-605 [8]
- ↑ Chen D, Zhao CM. Genetically engineered mice: a new paradigm to study gastric physiology. Curr Opin Gastroenterol. 2007 Nov;23(6):602-6 [9]
- ↑ Lacroix-Fralish ML, Ledoux JB, Mogil JS. The Pain Genes Database: An interactive web browser of pain-related transgenic knockout studies. Pain 2007 Sep;131(1-2):3.e1-4 [10]
- ↑ Takao K, Yamasaki N, Miyakawa T. Impact of brain-behavior phenotypying of genetically-engineered mice on research of neuropsychiatric disorders. Neurosci Res. 2007 Jun;58(2):124-32 [11]
- ↑ Fievet C, Fruchart JC, Staels B. Genetically-engineered animals as research models for atherosclerosis: their use for the characterization of PPAR agonists in the treatment of cardiometabolic disorders. Front Biosci. 2007 May 1;12:4132-56 [12]
Wikimedia foundation. 2010.