- Mononegavirales
-
Mononegavirales
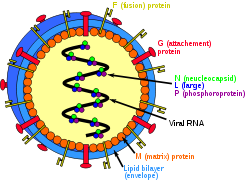
Estructura de HenipavirusClasificación de los virus Grupo: V (Virus ARN monocatenario negativo) Orden: Mononegavirales Familias Los Mononegavirales son un orden de virus comprendiendo especies que tienen un genoma de ARN no segmentado, de sentido negativo.
Contenido
Familias
- Bornaviridae (virus de la enfermedad de Borna)
- Rhabdoviridae (e.g. virus de la rabia)
- Filoviridae (e.g. virus Marburg, virus Ébola)
- Paramyxoviridae (e.g. virus de la enfermedad de Newcastle, virus del sarampión)
Un número importante de enfermedades humanas, (parotiditis, rabia) y las emergentes (fiebre hemorrágica ébola, enfermedad de borna, Hendra virus, (virus Nipah), son causadas por virus de este orden.
Estructura
Los viriones poseen una envoltura y un nucleocápsido helicoidal con una polimerasa RNA-dependiente (RDRP) y un ARN genómico. El genoma contiene 6-10 genes, aunque un proceso conocido como edición de ARN permite un gran número de productos génicos.
Ciclo de vida
Entrada
Las partículas virales entran a la célula por enlazarse a un receptor de célula superficial (e.g. ácido siálico) e induce la fusión entre la envoltura viral y la membrana celular. En el citoplasma, la partícula se descubre, lanzando el genoma.
Síntesis del mARN
La secuencia genómica es de sentido negativo, esto es que no codifica para proteínas. Las secuencias complementarias deben primero transcribirse con una polimerasa ARN dependIente (RDRP). Esto da inhabilidad al genoma para producir proteínas que requiere el virión en transportar el RDRP con él dentro de la célula. Esto es en contraste con las razas de virus positivas que pueden sintetizar RDRP una vez dentro de la célula.
El RDRP lanzado de la partícula viral se une al único promotor de secuencias al tercio final del genoma y comienza la transcripción. El RDRP pausa en los huecos entre cada gene, lanzando el mRNA completado. La traducción puede tanto terminar o continuar transcribiendo el siguiente gene. Esto crea una polaridad en la transcripción, donde los genes cierran el tercio final del genoma, transcribiendo en la máxima abundancia, mientras los del 5º final están al meos listos para ser transcriptos, desde el RDRP hay más oportunidades de terminar. Colocando los genes en orden de abundancia de mayor a menor, el virus rs capaz de usar esta polaridad como una forma de regulación transcripcional. En consecuencia, muchos genomas comienzan con el gene para la proteína del nucleocápsido y finalizan con el gene para RDRP.
Síntesis de proteína y de genoma
Once mRNA production has begun, the cellular protein translation system is co-opted to produce viral proteins which accumulate in the cytoplasm. At some point, possibly determined by the concentration of nucleocapsid protein, the RDRP molecules transcribing the viral genome begin ignoring the gap sequences between genes and produce full-length, positive-strand antigenomes. These are in turn transcribed into negative strand viral genome copies.
Ensamblaje y salida
The newly synthesised viral proteins and genomes self-assemble and accumulate near the inside of the cell membrane. The virions bud off from the cell, gaining an envelope from the cell membrane as they exit. The new viral particle infects another cell to repeat the cycle.
Editando el ARN
RNA editing is a mechanism used by some members of Mononegavirales to produce multiple proteins from a single gene. It occurs when the RDRP enzyme inserts extra residues into the mRNA being synthesised. This creates a frame-shift, altering the amino acid sequence encoded by the mRNA.
Evolución
As with other RNA viruses that do not have a DNA intermediate, members of Mononegavirales are able to evolve at a rapid rate due to the absence of proof-reading ability in the RDRP enzyme. A high mutation rate occurs in the production of new genomes (up to 1 per 1000 bases).
Enlaces externos
Referencias
 Wikimedia Commons alberga contenido multimedia sobre Mononegavirales. Commons
Wikimedia Commons alberga contenido multimedia sobre Mononegavirales. Commons Wikiespecies tiene un artículo sobre Mononegavirales. Wikispecies
Wikiespecies tiene un artículo sobre Mononegavirales. Wikispecies- Büchen-Osmond, C. (2003). 01. Mononegavirales. In: ICTVdB - The Universal Virus Database, version 3. Retrieved April 6, 2004 from http://www.ncbi.nlm.nih.gov/ICTVdb/ICTVdB/index.htm
- Dewhurst, S. (2003) University of Rochester Medical Center Department of Microbiology and Immunology Selected Virology Lecture Notes. Retrieved April 6, 2004 from http://www.urmc.rochester.edu/smd/mbi/grad2/pdf/gr03nns.pdf
Wikimedia foundation. 2010.
