- Pirosecuenciación
-
La pirosecuenciación o secuenciación 454, de 454 Life Sciences, es una tecnología de determinación de secuencia de DNA a gran escala, aplicable a genomas completos, mediante luminiscencia. A diferencia del sistema de Sanger, capaz de resolver 67.000 bases cada hora,[1] el método 454 puede determinar la secuencia de 20 millones en 4,5 horas, lo cual abarata enormemente el coste del proceso.[2]
Contenido
Fundamento
La muestra biológica se carga en una placa «PicoTiterPlate Device», de fibra óptica, que permite transmitir la luz producida por quimioluminiscencia durante la reacción de secuenciación hasta la cámara CCD de captación de imágenes. Dicha placa, en forma de panal, posee multitud de pocillos.
La particularidad es el tamaño del DNA a secuenciar en cada pocillo: debe ser entre a 500 y 1000 nucleótidos. La genoteca de DNA genómico con la cual comenzar la secuenciación se carga en la placa; en ella, cada fragmento se amplifica de forma aislada mediante emPCR o PCR basada en emulsión de modo que se amplifique hasta 10 millones de veces; este incremento facilita la detección de la señal.[2]
El fundamento de la secuenciación se basa en controlar el flujo secuencial y cíclico de nucleótidos sobre la placa en combinación con un sistema de detección bioluminiscente que permite convertir los productos generados durante la incorporación de nucleótidos (pirofosfato) en luz (por medio de la luciferasa) que es detectable por el dispositivo CCD.
Luminiscencia
La inyección de los dNTPs en cada pocillo acarrea la interacción con al menos un nucleótido complementario al situado en ese punto del DNA de secuencia desconocida; esto es que para cada punto de la secuencia sólo un dNTP puede ser individualmente unido de forma correcta, y cada vez que esto sucede el establecimiento de un enlace químico provoca la liberación de una molécula de pirofosfato.[1]
En cada pocillo de la placa PicoTiter se añade, además del DNA molde, la enzima luciferasa, que, en contacto con el pirofosfato, genera un haz de luz que indica la secuencia para ese punto; por último, no podemos olvidar el papel de la enzima apirasa, esta enzima es capaz de degradar aquellos nucleótidos que no se han unido en la reacción previa con el fin de que al añadir un nuevo dNTP, no existan restos de reacciones anteriores. [2]
Procedimiento
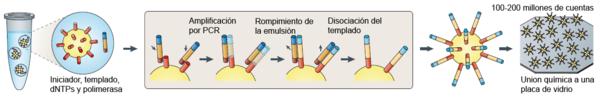 Pirosecuenciación 454. Esquema de sus fases.[3]
Pirosecuenciación 454. Esquema de sus fases.[3]
- Mediante una emulsión se generan microreactores (microesferas).
- En cada microreactor se encuentran los elementos necesarios para la reacción de PCR: iniciadores, dNTPs, polimerasa, cuentas de secuenciación y, por lo común, un único fragmento de ADN templado o molde.
- Mediante la amplificación por PCR, se generan muchas copias del mismo fragmento del templado original.
- Después se liberan las cuentas de la emulsión y se colocan en placas donde se procede con la secuenciación en paralelo.
Implicaciones
Su bajo coste y velocidad, que permitió a la compañía secuenciar el código de un adenovirus en 2003 en un sólo día,[1] plantea la posibilidad de secuenciar genomas completos de forma rutinaria, lo cual es de especial interés en biomedicina. De hecho, el genoma de James Watson, codescubridor de la estructura del DNA, fue descrito y publicado mediante esta técnica.[4]
Referencias
- ↑ a b c Cristina de Martos. «Más cerca de la secuenciación personalizada del ADN» (en español). Consultado el 15 diciembre de 2007.
- ↑ a b c «454 Life Sciences» (en inglés). Consultado el 15 diciembre de 2007.
- ↑ Metzker ML (2010) Sequencing technologies - the next generation. Nat Rev Genet 11: 31-46.
- ↑ «James Watson's Personal Genome Sequence» (en inglés).
Enlaces externos
Wikimedia foundation. 2010.
