- Marco abierto de lectura
-
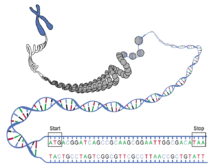 Esquema de un marco abierto de lectura, que incluye el codón de inicio (o start) y de parada (o stop).
Esquema de un marco abierto de lectura, que incluye el codón de inicio (o start) y de parada (o stop).
En genética se llama marco abierto de lectura (siglas ORF del inglés Open reading frame) a cada una de las secuencias de ADN comprendida entre un codón de inicio (AUG) de la traducción y un codón de terminación, descontando las secuencias que corresponden a los intrones en caso de haberlas. Se encuentra acotado por los UTRs, o secuencias no traducidas.
En una secuencia de ADN cualquiera hay, a priori, 6 posibles sentidos en los que pueden aparecer marcos abiertos de lectura; dado que cada codón toma 3 nucleótidos, existen 3 posibles lugares de inicio para tomar los nucleótidos de 3 en 3, si se tomara un cuarto nucleótido como lugar de inicio, haría coincidir el marco abierto de lectura con el mismo que si se toma el primer nucleótido. A lo que hay que sumar los otros 3 posibles marcos abiertos de lectura si el ADN es traducido tomando como molde la hebra complementaria, dando el sentido de lectura opuesto.
Estos marcos abiertos de lectura se denominan +1, +2, +3, -1, -2 y -3.
En un ejemplo con la secuencia 5' aactgcagtacgtaacgtca 3'
+3 5' a act gca gta cgt aac gtc a 3' +2 5' aa ctg cag tac gta acg tca 3' +1 5' aac tgc agt acg taa cgt ca 3' -1 3' ttg acg tca tgc att gca gt 5' -2 3' tt gac gtc atg cat tgc agt 5' -3 3' t tga cgt cat gca ttg cag t 5'
Cada uno de los 6 posibles marcos abiertos de lectura dará lugar a una secuencia proteica absolutamente diferente.
Véase también
- Codón
- Código genético
- Alineamiento de secuencias
- Bioinformática
- Predicción de genes
- Secuenciación de ADN
- UTR
Enlaces externos
- StarORF Un multi-plataforma, basada en Java, la herramienta de interfaz gráfica de usuario para predecir y analizar ORFs y la obtención de revertir secuencia del complemento.
Wikimedia foundation. 2010.
