- Ubiquitina
-
Ubiquitina HUGO 12468 Símbolo UBC Datos genéticos Locus Cr. 12 q24.3 Bases de datos Entrez 7315 OMIM 191340 RefSeq NM_021009 UniProt Q91888 La ubiquitina (o ubicuitina) es una pequeña proteína reguladora que ha sido encontrada en la mayoría de los tejidos de los organismos eucariotas. Una de sus muchas funciones es dirigir el reciclaje de proteínas. La ubiquitina puede asociarse a proteínas y marcarlas para su destrucción. El marcaje de ubiquitina dirige las proteínas al proteosoma, que es un gran complejo de proteínas que encontramos en la célula y que degrada y recicla proteínas innecesarias. Este descubrimiento ganó el premio Nobel en química en 2004.[1] [2]
Identificación
La ubiquitina (originalmente, polipéptido omnipresente –ubicuo- inmunopoyético) fue identificada en 1975 como una proteína de 8.5 kDa, de función desconocida, expresada en todas las células eucariotas. Las funciones básicas de la ubiquitina y los componentes de la vía de la ubiquitinización fueron aclaradas a principios de los años 1980 con el innovador trabajo realizado en el Fox Chase Cancer Center con Aaron Ciechanover, Avram Hershko, y Irwin Rose, por el cual recibieron el premio Nobel de Química en 2004.[1]
El sistema de ubiquitinización fue caracterizado al principio como un sistema proteolítico de ATP-dependencia presente en extractos celulares. Un polipéptido de calor estable presente en estos extractos, el factor 1 proteolítico ATP-dependiente (APF-1), fue encontrado en el proceso ATP (y Mg2+) dependiente de unión covalente del sustrato proteico a la lisoenzima. Diversas moléculas APF-1 se asocian a un solo sustrato molecular por un acoplamiento isopéptídico, y los conjugados que forman son rápidamente degradados con la liberación de APF-1. Poco después que la conjugación proteica APF-1 fuera caracterizada, la APF-1 fue identificada como ubiquitina. El grupo carboxilo (COOH) del residuo de glicina del carboneo terminal de ubiquitina (Gly76) fue identificado como la mitad conjugada al sustrato de los residuos de lisina.
La proteína
Propiedades de la ubiquitina (en humanos) Número de residuos 76 Masa molecular 8564.47 Da Punto isoeléctrico (pI) 6.79 Nombre de los genes RPS27A (UBA80, UBCEP1), UBA52 (UBCEP2), UBB, UBC La ubiquitina es una pequeña proteína que existe en todas las células eucariotas. Ésta realiza sus funciones gracias a la conjugación con una gran gama de proteínas diana. La ubiquitina está formada de 76 aminoácidos y tiene una masa molecular de aproximadamente 8.5 kDa. Entre sus características importantes se distinguen su cola C-terminal y los 7 residuos de lisina. Su estructura está sumamente conservada en el linaje eucariota: la ubiquitina del ser humano y la ubiquitina de la levadura comparten un 96 % de la secuencia identificadora de aminoácidos.
La secuencia humana de la ubiquitina, en el código de una letra, (los residuos de lisina, en negrita) es la siguiente:
MQIFVKTLTGKTITLEVEPSDTIENVKAKIQDKEGIPPDQQRLIFAGKQLEDGRTLSDYNIQKESTLHLVLRLRGG Los genes
En los mamíferos, la ubiquitina está codificada por 4 genes diferentes. El UBA52 y el RPS27A son los genes que codifican las copias de ubiquitina que se acoplan a las proteínas ribosómicas L40 y S27a (respectivamente) para su traducción. También encontramos el UBB y el UBC, que son codificadores de proteínas precursoras de la poliubiquitina.[3]
El origen
No se conoce ninguna ubiquitina ni la maquinaria de ubiquitinización necesaria en las células procariotas. Sin embargo, la ubiquitina se cree que desciende de proteínas procariotas similares a ThiS[4] o MoaD.[5] Estas proteínas procariotas, a pesar de tener poca secuencia de identificación (ThiS contiene únicamente un 14 % de secuencia identificadora respecto la ubiquitina), comparten la misma conformación proteica. Además comparten también química del azufre con la ubiquitina. Así, el MoaD, que está implicado en la biosíntesis del cofactor molibdeno, interacciona con el MoeB, que actúa como una enzima E1 de activación de la ubiquitina para MoaD, reforzando la asociación entre estas proteínas procariotas y el sistema de ubiquitina. Un sistema similar es el que existe para el ThiS, con su enzima E1 ThiF. Así, también se cree que la proteína Urm-1 de la bacteria Saccharomyces cerevisiae, que es un modificante asociado a la ubiquitina, es " un fósil molecular " que conecta la relación evolutiva entre las proteínas procariotas similares a la ubiquitina i dicho polipéptido.[6]
La ubiquitinización
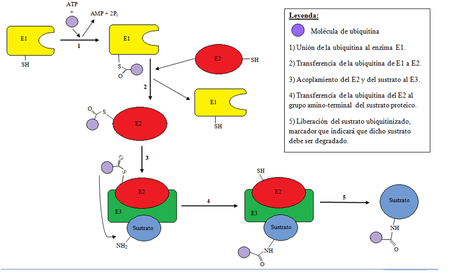
La ubiquitinización es un proceso enzimático de modificación proteica post-traduccional (PTM) en el cual el ácido carboxílico de la glicina terminal (que encontramos en el motivo de di-glicina de la ubiquitina activada) forma un enlace amida con el grupo amino épsilon de la lisina en la proteína modificada. El proceso de marcar una proteína con ubiquitina (ubiquitinización) consta de una serie de pasos:
- Activación de la ubiquitina: la ubiquitina es activada en una reacción de dos pasos por una enzima activadora de ubiquitina en un proceso que requiere ATP como fuente de energía.
El paso inicial implica la producción de un producto intermedio adenilil-ubiquitina. El segundo paso consiste en transferir la ubiquitina al centro activo del E1, concretamente uniéndose al residuo de cisteína, con la liberación de AMP. Este paso se realiza mediante un acoplamiento tioéster entre el carboneo terminal del grupo carboxilo de la ubiquitina i el grupo sulfhídrico de la cisteína del E1.
- Transferencia de la ubiquitina del centro activo del E1 a una enzima E2 de conjugación de ubiquitina a través de una reacción de transtioesterificación. Los genomas de los mamíferos contienen entre 30 y 40 UBCs.
- El paso final de la cascada de ubiquitinización crea un enlace isopeptídico entre una lisina de la proteína diana y la glicina del carboneo terminal de la ubiquitina. En general, este paso requiere la actividad de una de los cientos de enzimas ligasas E3 de ubiquitina (a menudo llamada simplemente ligasa de ubiquitina). Las enzimas E3 funcionan como los centros de reconocimiento del sustrato del sistema y son capaces de interaccionar tanto con el E2 como con el sustrato.
En la cascada de ubiquitinización, la enzima E1 puede unirse con docenas de enzimas E2, que pueden a su vez unirse con unos cientos de enzimas E3 de un modo jerárquico. Otras proteínas similares a la ubiquitina (como las ULPs) también se modifican a través de la cascada E1-E2-E3.
En la imagen podemos ver gráficamente el proceso de ubiquitinización descrito.
E3
Las enzimas E3 poseen uno de los siguientes posibles dominios:
- El dominio HECT (Homólogo al E6-AP Carboxilo Terminal)
- El dominio RING (Nuevo Gen Realmente Interesante) o el tan estrechamente relacionado dominio U-box
Así, la transferencia e interacción puede ocurrir de dos modos:
- Directamente de la enzima E2, catalizada por el dominio RING.
- Vía una enzima E3, catalizada por el dominio HECT. En este caso se forma un intermediario con la unión covalente entre la E3 y la ubiquitina, antes de la trasferencia de la ubiquitina a la proteína sustrato.
El complejo de promoción de la anafase (APC) y el complejo SCF (por Skp1-Cullin-F-complejo proteico de caja –o box-) son dos ejemplos de subunidades de las E3 envueltas en el proceso de reconocimiento y ubiquitinización de proteínas diana específicas para su degradación en el proteosoma.
Función y modificación de las distintas ubiquitinas
Después de la adición de una sola ubiquitina a un sustrato proteico (monoubiquitinización), más moléculas de ubiquitina pueden ser añadidas a la proteína en cuestión, produciendo una cadena proteica poliubiquitinizada. Además, hay que destacar que algunos sustratos son modificados con la adición de dichas moléculas de ubiquitina a los múltiples residuos de lisina de la proteína diana en un proceso llamado multiubiquitinización.
La ubiquitina posee un total de 7 residuos de lisina. En un principio los tipos de cadenas de ubiquitina identificadas eran aquellos unidos por vía lisina-48. Sin embargo, un trabajo más reciente ha dado a conocer una amplia variedad de acoplamientos que implican a todos los residuos posibles de lisina[7] [8] y además cadenas unidas al extremo amino terminal de una ubiquitina (también llamadas “cadenas lineales”).[9] El trabajo, publicado en 2007, ha demostrado la formación de cadenas de ubiquitina con bifurcaciones que contienen múltiples tipos de unión.[10] Hay que destacar además que se han encontrado cadenas de ubiquitina "atípicas" (sin unión a lisina-48) y han sido publicadas en una revista por Ikeda y Dikic.[11]
El sistema de ubiquitinización funciona en una gran variedad de procesos celulares, incluyedo:[12]
Procesación de antígenos
- Apoptosis
- Biogénesis de las organelas
- Ciclo celular y división
- Transcripción y reparación del ADN
- Diferenciación y desarrollo
- Respuesta inmunológica e inflamación
- Degeneración muscular y neuronal
- Morfogénesis de las redes neuronales
- Modulación de los receptores de la superficie celular, canales iónicos y vías de secreción
- Respuesta al estrés y a moduladores extracelulares
- Biogénesis ribosómica
- Infección viral
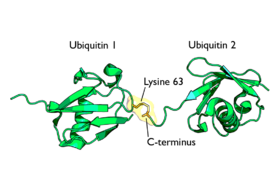
Cadenas vinculadas a lisina
La cadenas poliubiquitinizadas más estudiadas (las unidas a lisina-48) marcan proteínas para su posterior destrucción, proceso llamado proteólisis. Al menos cuatro moléculas de ubiquitina tienen que estar unidas a residuos de lisina de la proteína a destruir para que ésta sea reconocida por la proteína 26S. Las cadenas unidas a la lisina 63 dirigen la localización de las proteínas. La monoubiquitinización de las proteínas también marca la localización de las proteínas. El proteasoma o proteosoma[13] es un complejo, con estructura en forma de barril y dos cámaras en su interior, donde la proteólisis tiene lugar. Las proteínas son degradadas rápidamente en pequeños péptidos (normalmente de 3 a 24 residuos aminoacídicos de longitud). Las moléculas de ubiquitina se escinden de la proteína inmediatamente antes de la destrucción y son recicladas para un uso posterior. Aunque la mayoría de los substratos que van a degradarse al proteasoma son ubiquitinizados, existen algunas proteínas que no son ubiquitinizadas y que sin embargo se destinan también al proteasoma.
Diagrama de diubiquitina ligada a la lisina-48. La unión (mostrada en naranja) tiene lugar entre las dos cadenas de ubiquitina.Diagrama de diubiquitina ligada a lisina-63. La unión (como se muestra en naranja) tiene lugar entre las dos cadenas de ubiquitina.Monoubiquitinización
La ubiquitina puede marcar también a proteínas transmembrana (por ejemplo a los receptores) para su extracción desde las membranas y cumplir varios papeles de señalización dentro de la célula. Las moléculas transmembrana de la superficie celular que están marcadas con ubiquitina están a menudo monoubiquitinizadas, y esta modificación altera la localización subcelular de la proteína, marcando normalmente la proteína para la destrucción en los lisosomas.
Las histonas estan normalmente monoubiquitinizadas y asociadas con la señalización o el marcaje estructural.
Otros tipos de cadenas
La ubiquitina tiene siete residuos de lisina que pueden servir como puntos de poliubiquitinización, que son: K48, K63, K6, K11, K27, K29 y K33. Estos puntos de unión pueden definir y determinar señales únicas que son reconocidas por proteínas de unión a la ubiquitina, las cuales tienen motivos de interacción con la ubiquitina (UIM) con los que se unen a la propia molécula proteica. Se cree que las distintas uniones son reconocidas por proteínas que son específicas para reconocer la topología intrínseca del enlace. Un ejemplo es la unión K63, conocida por estar implicada en el reconocimiento del ADN dañado en zonas de rotura de la doble cadena helicoidal del material hereditario. La unión K63 parece estar colocada en la histona H2AX por el par de ligasas E2/E3, Ubc13-Mms2/RNF168. Esta cadena de K63 aparece para elegir RAP80, que contiene UIM, y así RAP80 ayuda a localizar BRCA1. Esta vía elegirá las proteínas necesarias para la reparación de la recombinación homóloga.
Enfermedades asociadas
Trastornos genéticos
Algunos trastornos genéticos asociados a la ubiquitina son:
- El gen que controla la ubiquitina-1 se puede encontrar en lesiones asociadas con el Alzheimer y el Parkinson. Dos variantes de la transcripción que codifican diferentes isoformas se han encontrado para este gen. Elevados niveles de ubiquitina en el cerebro disminuyen la malformación de la molécula APP, que juega un papel clave en la aparición del Alzheimer.[14] Conversly, lower levels of ubiquilin-1 in the brain were associated with increased malformation of APP.[14] Por el contrario, bajos niveles de ubiquitina-1 en el cerebro se asocian con un aumento de las malformaciones de APP.[14]
- El gen cuyo trastorno causa el Síndrome de Angelman, el UBE3A, codifica a la ubiquitina ligasa (E3), enzima llamada E6-AP
- El gen mutado en el Síndrome de Von Hippel-Lindau codifica a la ubiquitina E3 ligasa llamada el supresor de tumores VHL o el gen VHL
- El gen mutado en el Síndrome de Liddle causa una desregulación de un canal epitelial de Na+ (ENaC) y en consecuencia, causa hipertensión
- Ocho de los trece genes identificados cuya mutación causa la anemia Fanconi codifican proteínas que forman un complejo de ubiquitina ligasa (E3) largo.
- Mutaciones de la Cullin 7 E3 ubiquitina ligasa están relacionadas con el Síndrome 3-M, un trastorno autosómico recesivo que causa retraso del crecimiento.[15]
Mutación con desplazamiento en la pauta de lectura (UBB+1)
El gen de la ubiquitina B puede estar transcrito incorrectamente debido a una secuencia monótona de nucleótidos en la región codificante del gen. Como resultado, se produce una eliminación de un dinucleótido en el ARNm causando un desplazamiento en la pauta de lectura. Cuando se traducen a proteínas, las ubiquitinas disfuncionales han perdido su C-terminal de glicina y en su lugar tienen un péptido de 20 aminoácidos (Ubiquitina B+1 o UBB+1). Se ha demostrado que las UBB+1 se acumulan de forma selectiva en tautopatías y poliglutaminopatías.
Inmunoquímica
Los anticuerpos anti-ubiquitina son usados en histología para identificar acumulaciones anormales de proteína dentro de las células que son marcadores de la enfermedad. Estas acumulaciones se llaman cuerpos de inclusión (inclusión bodies). Algunos ejemplos de estas inclusiones anormales en las células son
- Ovillos neurofibrilares en el Alzheimer
- Cuerpos de Lewy en el Parkinson
- Cuerpos de Pick en la enfermedad de Pick
- Inclusiones en la enfermedad de la motoneurona y la enfermedad de Huntington
- Cuerpos de Mallory en la hepatopatía alcohólica
- Fibras de Rosenthal en los astrocitos
Modificadores parecidos a la ubiquitina
Aunque la ubiquitina es el modificador post-traduccional mejor entendido, existe una familia creciente de proteínas similares a las ubiquitinas (UBL) que modifican las dianas celulares de una manera paralela y a la vez distinta a la de la ubiquitina. Las UBL conocidas incluyen: pequeños modificadores parecidos a la ubiquitina (SUMO), ubiquitina de reacción cruzada (UCRP, también conocidas como gen estimulado por interferón-15 ISG15), ubiquitina relacionada con el modificador-1 (URM1), proteína-8 reguladora del desarrollo y expresión de la célula neuronal precursora (NEDD8, también llamada Rub1 en el S. cerevisiae), antígeno leucocitario humano F-asociado (FAT10), autofagia-8 (ATG8) y -12 (ATG12), Fau ubiquitina (FUB1), MUB (UBL anclada a la membrana),[16] ubiquitina dobladora modificadora-1 (UFM1) y proteína similar a la ubiquitina-5 (UBL5, que es más conocida como homóloga a la ubiquitina-1 (Hub-1) en el S. pombe).[17] [18] Si bien estas proteínas sólo comparten la secuencia identificadora primaria con la ubiquitina, están estrechamente relacionadas tridimensionalmente. Por ejemplo, las SUMO sólo comparten un 18% de secuencia identidad respecto la ubiquitina, pero contienen el mismo pliegue estructural. Este pliegue se llama “pliegue de la ubiquitina” o a veces pliegue ubiquitón. FAT 10 y UCRP contienen dos de estos pliegues. Este compacto pliegue beta se encuentra en la ubiquitina, en las UBL y en las proteínas que comprenden un dominio de ubiquitina.
Estas moléculas relacionadas entre sí tienen nuevas funciones e influencia en diversos procesos biológicos. También hay una regulación cruzada entre varias de las vías de conjugación dado que algunas proteínas pueden ser modificadas por más de una UBL, y a veces incluso en el mismo residuo de lisina. Por ejemplo, modificaciones en las SUMO a menudo actúan de forma antagónica a la ubiquitinización y sirven para estabilizar substratos proteicos. Proteínas conjugadas con las UBL no son identificadas para degradarse en el proteasoma, sino que tienen función en diversas actividades reguladoras. La unión de las UBL puede alterar la conformación del substrato, afectar a la afinidad por los ligandos u otras moléculas de interacción, alterar la localización del substrato e influenciar en la estabilidad de la proteína. Las UBL son estructuralmente similares a la ubiquitina y son procesadas, activadas, conjugadas y liberadas del conjugado por pasos enzimáticos parecidos a los correspondientes de la ubiquitina. Las UBL son también traducidas con extensiones C-terminal que son procesadas para exponer la invariante C-terminal LRGG. Estos modificadores tienen sus propias enzimas específicas E1 (activación), E2 (conjugación) y E3 (ligación) que conjugan las UBL a dianas intracelulares. Estos conjugados pueden ser revertidos por unas isopeptidasas UBL específicas que tienen mecanismos similares a los de las enzimas de desubiquitinización.[12]
Dentro de algunas especies, el reconocimiento y destrucción de las mitocondrias de los espermatozoides tiene lugar a través de un mecanismo en el que la ubiquitina está implicada, siendo éste el responsable de la eliminación de las mitocondrias después de la fertilización.[19]
Proteínas humanas que contienen dominios de ubiquitina
ANUBL1; BAG1; BAT3; DDI1; DDI2; FAU; HERPUD1; HERPUD2; HOPS; IKBKB; ISG15; LOC391257; MIDN; NEDD8; OASL; PARK2; RAD23A; RAD23B; RPS27A; SACS;SF3A1; SUMO1; SUMO2; SUMO3; SUMO4; TMUB1; TMUB2; UBA52; UBB; UBC; UBD; UBFD1; UBL4; UBL4A; UBL4B; UBL7; UBLCP1; UBQLN1; UBQLN2; UBQLN3; UBQLN4; UBQLNL; UBTD1;UBTD2; UHRF1; UHRF2;
Véase también
Referencias
- ↑ a b «The Nobel Prize in Chemistry 2004». Nobelprize.org. Consultado el 16-10-2010.
- ↑ «The Nobel Prize in Chemistry 2004: Popular Information». Nobelprize.org. Consultado el 16-10-2010.
- ↑ Kimura Y, Tanaka K (2010). «Regulatory mechanisms involved in the control of ubiquitin homeostasis». J Biochem 147 (6): pp. 793–8. doi:. PMID 20418328.
- ↑ «Solution structure if ThiS and implications for the evolutionary roots of ubiquitin». Nature Structural Biology 8 (1): pp. 47–51. 2001. doi:. PMID 11135670. http://www.nature.com/nsmb/journal/v8/n1/full/nsb0101_47.html.
- ↑ «Mechanism of ubiquitin activation revealed by the structure of a bacterial MoeB–MoaD complex». Nature 414 (6861): pp. 325–329. 2001. doi:. PMID 11713534. http://www.nature.com/nature/journal/v414/n6861/abs/414325a0.html.
- ↑ «Origin and Function of Ubiquitin-like Protein Conjugation». Nature 458 (7237): pp. 422–429. 2009. doi:. PMID 19325621. PMC 2819001. http://www.nature.com/nature/journal/v458/n7237/full/nature07958.html.
- ↑ «Characterization of Polyubiquitin Chain Structure by Middle-down Mass Spectrometry». Analytical chemistry 80 (9): pp. 3438–44. May 2008. doi:. ISSN 0003-2700. PMID 18351785.
- ↑ «A proteomics approach to understanding protein ubiquitination». Nature biotechnology 21 (8): pp. 921–6. Aug 2003. doi:. ISSN 1087-0156. PMID 12872131.
- ↑ «A ubiquitin ligase complex assembles linear polyubiquitin chains» (Free full text). The EMBO journal 25 (20): pp. 4877–87. Oct 2006. doi:. ISSN 0261-4189. PMID 17006537.
- ↑ «Certain pairs of ubiquitin-conjugating enzymes (E2s) and ubiquitin-protein ligases (E3s) synthesize nondegradable forked ubiquitin chains containing all possible isopeptide linkages» (Free full text). The Journal of biological chemistry 282 (24): pp. 17375–86. Jun 2007. doi:. ISSN 0021-9258. PMID 17426036. http://www.jbc.org/cgi/pmidlookup?view=long&pmid=17426036.
- ↑ «Atypical ubiquitin chains: new molecular signals. 'Protein Modifications: Beyond the Usual Suspects' Review Series» (Free full text). EMBO reports 9 (6): pp. 536–42. Jun 2008. doi:. ISSN 1469-221X. PMID 18516089.
- ↑ a b «Ubiquitin Proteasome Pathway Overview». Archivado desde el original, el 2008-03-30. Consultado el 30-04-2008.
- ↑ «Recognition of the polyubiquitin proteolytic signal» (Free full text). The EMBO journal 19 (1): pp. 94–102. Jan 2000. doi:. ISSN 0261-4189. PMID 10619848.
- ↑ a b c Stieren ES, El Ayadi A, Xiao Y, Siller E, Landsverk ML, Oberhauser AF, Barral JM, Boehning D (August 2011). «Ubiquilin-1 Is a Molecular Chaperone for the Amyloid Precursor Protein». J Biol Chem 286 (41): pp. 35689–98. doi:. PMID 21852239. Resumen divulgativo – Science Daily.
- ↑ «Identification of mutations in CUL7 in 3-M syndrome». Nature genetics 37 (10): pp. 1119–24. Oct 2005. doi:. ISSN 1061-4036. PMID 16142236.
- ↑ «MUBs, a Family of Ubiquitin-fold Proteins That Are Plasma Membrane-anchored by Prenylation». Journal of Biological Chemistry 281 (37): pp. 27145–27157. July 2006. doi:. ISSN 0021-9258. PMID 16831869.
- ↑ Welchman RL, Gordon C, Mayer RJ (2005). «Ubiquitin and ubiquitin-like proteins as multifunctional signals». Nat Rev Mol Cell Biol 6 (8): pp. 599–609. doi:. PMID 16064136.
- ↑ Grabbe C, Dikic I (2009). «Functional roles of ubiquitin-like domain (ULD) and ubiquitin-binding domain (UBD) containing proteins». Chem Rev 109 (4): pp. 1481–94. doi:. PMID 19253967.
- ↑ Ubiquitinated sperm mitochondria, selective proteolysis, and the regulation of mitochondrial inheritance in mammalian embryos. Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G.
Enlaces externos
Académicos
- UniProt entry for ubiquitin
- UbiPred — server for the prediction of ubiquitylation sites
- Ubiquitin Web-page
- 7.340 Ubiquitination: The Proteasome and Human Disease MIT OpenCourseWare. Notes from MIT course.
Comerciales
- Enzo Life Sciences : Ubiquitin and Proteasome Research Products
- Boston Biochem : Ubiquitin and Ubiquitin-like Research Products
- Progenra Inc. : Ubiquitin-based Biotechnology Corporation
Categorías:- Genes del cromosoma 12
- Proteínas
Wikimedia foundation. 2010.