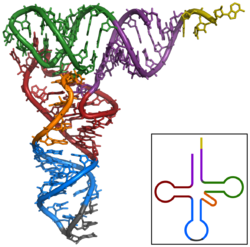
- ARN no codificante
-
Un ARN no codificante (ncARN) es una molécula de ARN funcional que no se traduce en una proteína. Con menos frecuencia son usados sinónimos como npcARN (non-protein-coding ARN), nmARN (non-messenger ARN), snm ARN (small non-messenger ARN) y fARN (functional ARN). El termino small ARN (sARN). Se utiliza comúnmente para las pequeñas ncRNAs de bacterias . La secuencia de ADN desde la cual es trascrito el ncARN como producto final a menudo se denomina RNA gene o non-coding RNA gene
Los genes de ARN no codificante incluyen ARNs abundantes e importantes funcionalmente, como el ARN transferent (tARN) y ARN ribosòmic (rARN), así como ARNs como el snoARNs, microARNs, siARNs y piARNs y el ncARN largo que incluye ejemplos como el Xist y HOTAIR. El número de ncARNs codificantes en el genoma humano es desconocido; sin embargo, recientes estudios transcriptomicos y bioinformáticos sugieren la existencia de miles de ncARNs. [1] [2] [3] [4] Como la función de muchos de los ncARNs identificados recientemente no esta confirmada, es posible que muchos sean no funcionales. [5][1]
Contenido
Historia y Descubrimiento
Plantilla:Further Los ácidos nucleicos fueron descubiertos por primera vez en 1868 por Friedrich Miescher [6] y en 1939 ya se había implicado el ARN a la síntesis de proteínas.[7] Décadas mas tarde, Francis Crick va a predecir un componente de el ARN funcional que controlaba la traducción, argumentaba que el ARN encajaba mejor en el trascrito de mARN que en un polipéptido puro.[8]</ref>
El primer ARN no codificante en ser caracterizado fue un tARN de alanina la cual se hallaba en la levadura utilizada para pastelería, su estructura se publicó en 1965.[9] Para producir una muestra (purificada) de tARN de alanina, Robert W. Holleyet al. utilizo 140 Kg. de levadura de pastelería comercial para obtener no mas de 1 g de tARNA purificada para su análisis.[10] El tARN de 80 nucleótidos fue secuenciado primero digerida en Ribonucleasa pancreática (producto fragments que acaban en Citosina o Uridina) y después en ribonucleasa takadiastasa Tl (producto fragments que acaba en Guanosina). La cromatografía y la identificación de los extremos 5' y 3' va ayudar después a ordenar los fragments para estabilizar la secuencia de ARN.[10] De las tres estructuras propuestas originalmente para este tARN,[9] la estructura en forma de trébol va a ser propuesta de forma independiente en siguientes publicaciones.[11][12][13][14] La estructura secundaria en forma de trébol va ser concretada después de un análisis con cristalografía de rayos X realizado por dos grupos de investigación independientes en 1974
El ARN ribosomal fue el siguiente en ser descubierto, seguido por el UARN a principios de los años 80. Desde entonces el descubrimiento de nuevos ARNs no codificantes a continuado con los snoARNs, Xist, CRISPR y muchos mas.[17] Adiciones notables recientes incluyen Riboswitches y el miARN, el descubrimiento del mecanismo de el ARNi fue descubierto por Craig C. Mello y Andrew Fire (ganadores en el 2006 del Premio Nóbel de Medicina y Fisiología)
Funciones Biológicas del ncARN
Los ARNs no codificantes pertenecen a diversos grupos y participan en muchos procesos celulares. Estos van desde los ncARNs de vital importancia que se conservan en todos o la mayoría de la vida celular a través de ncARNs mas transitorios, que son específicos pera una o unas pocas especies relacionadas. Se cree que los ncARNs mas conservados son fósiles o reliquias moleculares de LUCA y el mundo del ARN.[19][20][21]
Traducción en ncRNAs
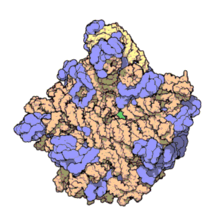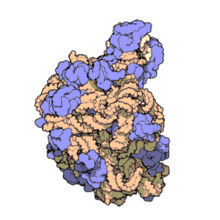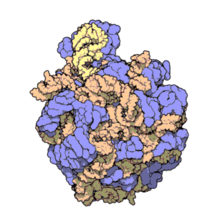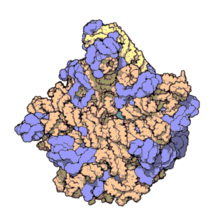 Atomic structure of the 50S Subunit from Haloarcula marismortui. Proteins are shown in blue and the two RNA strands in orange and yellow.[2] The small patch of green in the center of the subunit is the active site.
Atomic structure of the 50S Subunit from Haloarcula marismortui. Proteins are shown in blue and the two RNA strands in orange and yellow.[2] The small patch of green in the center of the subunit is the active site.
Muchos de los ncARNs esenciales y abundantes conservados están implicados en la traducción. Las partículas de ribonucleoproteina (RNP) son llamadas "fabricas" donde la traducción tiene lugar en la célula. El ribosoma cuenta con mas del 60% de ARN ribosomal, formado por 3 ncARNs en procariotas y 4 ncARNs en eucariotas. Los ARNs ribosomales catalizan la traducción de secuencias de nucleótidos a proteína. Otro tipo de ncARNs, los ARNs de transferencia, forman una “molécula adaptadora” entre el mARN y las proteínas. Los snoARNs H/ACA box i C/D box son ncARNs que se encuentran en archaea y eucariotas y la ARNasa MRP esta restringida a solo a los eucariontes, ambos grupos de ncARN están implicados en la maduración del rARN. Los snoARNs guían modificaciones covalentes de rARN, tARN i snARNs, la ARNasa MRP rompe el espaciador 1 trascrito interno entre 18S u 5,8S rARNs. El ncARN omnipresente, ARNasa P, es un pariente evolutivo de la ARNasa MRP.[23] la ARNasa P madura secuencias de tARN generando extremos 5' maduros de tARN y a través de escindir los elementos líderes 5' de los tARNs precursores. Otro omnipresente RNP llamada SRP reconoce y transporta nuevas proteínas al retículo endoplasmatico en eucariotas y a la membrana plasmática en procariotas. En bacterias, el ARN Transfer-messenger RNA (tmRNA)N) es un RNP implicado en rescatar ribosomas estancados, etiquetando polipéptidos incompletos y promoviendo la degradación de mARN en mal estado.
ncRNAs in RNA splicing (empalme)
En los eucariotas la spliceosome realiza las reacciones de empalme, escenciales para retirar las secuencias de los Intrones. Este proceso es necesario pera la formación de mARN maduro. El spliceosoma es un empalmador RNP a menudo también conocido como snRNP o tri-snRNP. Hay dos formas diferentas de spliceosoma, la mayor y la menor. Los componentes de ncARN del spliceosoma mayor son U1, U2, U3, U4 i U5. Los componentes de ncARN del spliceosoma menor son U11, U12, U5, U4atac i U6atac.
Otro grupo de intrones puede catalizar su propia eliminación de las transcripciones acogidas. Estos son llamados self-splicing ARNs y se dividen en dos grupos principales, el grupo I de intrones catalíticos y el grupo II de intrones catalíticos. Estos ncARNs catalizan su propia escisión a partir de los precursores de mARN, tARN i rARN en una gran diversidad de organismos
En mamíferos, se ha descubierto que los snoARNs pueden regular el splicing alternativo del ARNm. Por ejemplo, el snoARN HBII-52 regula el splicing del receptor de serotonina 2C.[24] Kishore S, Stamm S (2006). «The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C». Science 311 (5758): pp. 230–231. doi:. PMID 16357227.</ref>
En los nematodos, el SmY ncARN parece estar involucrada en el trans-splicing del mARN.
ncARNs en la Regulación Genética
La expresión de muchos miles de genes son regulados por ncRNAs. Esta regulación se puede producir en el transporte o en cis.
trans-acting ncRNAs
En eucariotas superiores los microARNs regulan la expresión genética. Un solo miARN puede reducir los niveles de expresión de centenares de genes. El mecanismo por el cual las moléculas de miARN maduro actúan a través de moléculas parcialmente complementarias a uno o mas mARNs, generalmente en 3'UTRs. La función principal de los miARNs es inhibir la expresión genética.
Se ha demostrado que la ncARN ARNasa P también influye sobre la expresión genética. En el núcleo humano, la ARNasa P es necesaria para la trascripción normal y eficiente de diversos ncARNs transcritos por el ARN polimerasa III. Estos incluyen genes de tARN, 5S rARN, SRP ARN i U6 snARN. La ARNasa P ejerce su función en la trascripción asociándose con la Pol III y la cromatina de los genes actúan de tARN y 5S rARN.[25] [3]
Se ha demostrado que el 7SK ARN, un ncARN metazoal, actúa como un regulador negativo del factor de elongación P-TEFb de la ARN polimerasa II, y que esta actividad está influenciada por las vías de respuesta al estrés.
El ncARN bacterial, 6S ARN, se asocia específicamente en la holoenzima ARN polimerasa, que cuenta con el factor específico sigma 70. Esta interacción reprime la expresión de un promotor dependiente de sigma 70 durante la fase estacionaria.
Otro ncARN bacterial, el OxyS ARN, reprime la traducción asociándose a secuencias de Shine-Dalgarno obstaculizando la unión de los ribosomas. El OxyS ARN es producido como respuesta al estrés oxidativo en Escherichia coli.
El B2 ARN es un pequeño trascrito de ncARN polimerasa III que reprime la trascripción del mARN en respuesta al choque térmico producido en las células del ratón. El B2 ARN inhibe la trascripción asociándose al núcleo de la Pol II. A través de esta interacción, el B2 ARN reúne complejos de preiniciación en el promotor y bloquea la síntesis de ARN.[26] [4]
Un estudio reciente ha demostrado que solo el acto de trascripción de la secuencia de ncARN puede tener influencia sobre la expresión genética. La trascripción a ncARN de la ARN polimerasa II se necesita para la remodelación de la cromatina en los chizosaccharomyces pombe. La cromatina adopta progresivamente una configuración abierta, así es como diversas especies de ncARN son transcritos.[27] [5]
cis-acting ncRNAs
Un número de ncARNs están unidos a los 5'UTRs de los genes codificantes de proteínas e influencian su expresión de diversas maneras. Por ejemplo, un riboswitch puede unir directamente una molécula objetivo pequeña y esta unión afecta la actividad genética.
Se encuentran secuencias de ARN líder en la zona superior del primer gen de los peronés biosintetitzadores de aminoácidos. Estos elementos de ARN forman una de les dos posibles estructures en regiones codificadas con secuencias péptidas muy cortas que son ricos en aminoácidos producto final del operon. Una estructura degradadota se forma cuando existe un exceso de los aminoácidos reguladores y el movimiento ribosómico sobre la trascripción del líder no se simplifica. Cuando hay ha una deficiencia de tARN cargado de el aminoácido regulador, el ribosoma actúa formando la estructura no-degradativa. Así permite al ARN polimerasa transcribir el operón. Los ARN líderes conocidos son el operó líder de histidina, el operón líder de leucina, el operón líder de treonina y el operón líder de triptòfan.
El IRE (“Iron reponse elements”, Elementos de respuesta al hierro) están unidos por IRP (Iron response proteins”, proteínas de respuesta al hierro). El IRE se encuentra en UTRs de diferentes mARNs diferentes producidos que participan en el metabolismo del hierro. Cuando la concentración del hierro es baja, los IRPs conducen la IRE mARN de ferritina para la represión de su traducción.
IRES (“Internal ribosome entry sites”, sitios de entrada internos de los ribosomas) són una estructura de ARN que permiten la iniciación de la traducción en la mitad de la secuencia de mARN como parte del proceso de síntesis proteica.
ncRNAs y la defensa del Genoma
Los piARNs (“Piwi-interacting ARNs”, ARN que interactúan en el Piwi) expresados en los testículos de los mamíferos y las células somáticas, forman complejas proteínas de ARN en proteínas Piwi. Estas complejas piARNs están relacionadas a la inhibición de la trascripción genética de los retrotransposones y otros elementos genéticos de las células de la línea germinal, especialmente aquellas implicadas en la espermatogénesis
El CRISPR (“Clustered Regularly Interspaced Short Palindromic Repeats”) son repeticiones encontradas en el DNA de muchas bacterias y de la archea. Las repeticiones están separadas por espacios de longitud similar. Se ha estado demostrando que estos espacios pueden derivar de infecciones víricas y, consecuentemente, pueden ayudar a las células a protegerse de las infecciones.
ncRNAs y la estructura Cromosomica
La telomerasa es una enzima RNP que añade repeticiones de secuencias específicas de DNA ("TTAGGG" en vertebrados) a regiones teloméricas, las cuales se encuentran al final de los cromosomas de los eucariotas. Los telomeros contienen DNA condensado, dando estabilidad a los cromosomas. La enzima es una transcriptaza inversa que lleva ARN telomerasa, que es utilizada para alargar la telòmerasa, que se acorta después de cada ciclo de replicación.Xist (“X-inactive-specific transcript”, trascripción inactivo especifico de X) es un gen largo ncARN de los cromosomas X de los mamíferos placentarios que actúa como un efector de la inactivación del proceso de formación de cuerpos Barr por el cromosoma X. Un antisense RNA, Tsix, es un regulador negativo de Xist. Los cromosomas X con falta de expresión de Tsix ( por lo tanto tiene un alto nivel de trascripción de Xist) son inactivados mas frecuentemente que en los cromosomas normales. En los drosofilidos, que también utilizan un sistema de determinación del sexo XY, Los ARNs roX (ARN del cromosoma X) están involucrados en la compensación de la dosis.[28] Tanto Xist como roX presentan una regulación epigenètica de la trascripción a través de la captación de enzimas modificadoras de los histones.
RNA Bifuncional
Los ARNs bifuncionales son ARNs que tienen dos funciones diferentes.[29][30] También son conocidos como ARNs de doble función. La mayoría de los ARNs bifuncionales conocidos son mARNs que codifican una proteína y ncARNs. Sin embargo, también hay un creciente numero de ncARNs se dividen en dos categorías diferentes de ncARN. Por ejemplo, H/ACA box snoARN i miARN.[31][32][6] [7]
Dos ejemplos bien conocidos de ARNs bifuncionales son el SgrS ARN y el ARNIII. Sin embargo, se conocen también otros ARNs bifuncionales que existen: SRA,[33] VegT ARN,[34][35] Oskar ARN [36] i enod 40.[37][8] VegT RNA,[9] [10] Oskar RNA [11] and ENOD40.[12]
ncRNAs y las Enfermedades
Lease también: Long noncoding RNAs in disease
Proteínas, mutaciones y desequilibrios en el repertorio del ncARNs pueden producir una gran variedad de enfermedades.
Cancer
Muchos ncARNs muestran unos patrones de expresión anormales en los tejidos cancerosos. Estos incluyen miARNs,[38] mARN-like ncARNs largos, [39][40] GAS5[41] SNORD 50,[42]ARN telomerasa y YARNs.[43] Los miARNs están involucrados a gran escala regulatoria de muchos genes codificadores de proteínas;[44][45] Los YARN son importantes para la iniciación de la replicación de DNA[46] El ARN telonerazas que funciona como inicio para la telomeraza, un RNP que alarga las regiones telomèricas del final del cromosoma. La función directa del mARN-like ncARNs lago es muy clara.
Se ha demostrado que las mutaciones de la línea germinal en los precursores primarios del miR-16-1 y del miR-15 son mas frecuentes en los pacientes con leucemia linfoide crónica que en la población controlada.[47][48]
Se ha sugerido que un SNP extraño (rs11614913) que esta superpuesto a la hsa-mir-196a2 esta asociado al carcinoma pulmonar de las células pequeñas.[49] Del mismo modo, una pantalla de 17 miARNs que se ha previsto que regulen un numero de variaciones de genes asociados al cáncer de mama presentara variaciones al miR17 y miR-30c-1. Estos pacientes no eran portadores de de mutaciones BRCA1 ni BRCA2, arrojando la posibilidad de que el cáncer de mama hereditario pudiera ser causado por variaciones de estos miARNs.[50]
Síndrome Prader-Willi
La eliminación de las 48 copias del C/D box snoARN SNORD116 a estado demostrando como la primera causa del síndrome de Parder-Willi.[51][52][53] Prader-Willi es un desorden en el desarrollo asociado a problemas de comer en exceso y desordenes alimentarios. SNORD116 tiene sitios potenciales entre un número de genes codificadores de proteínas y podría tener un papel en la regulación alternativa del splicing.[54]
Autismo
El lugar cromosomal con que cuenta el grupo genético SNORD115 snARN ha estado duplicándose en aproximadamente un 5% de los individuos con características autista.[55] [56] Un ratón modificado genéticamente puede tener la duplicación del grupo SNORD115 manifiesta comportamiento autista.[57] [13]
Cartilage-hair hypoplasia
Se ha demostrado que mutaciones en ARNses MRP causa Cartilage-hair hypoplasia, una enfermedad asociada a un grupo de síntomas tales como baja estatura, escasez de cabello, anormalidades esqueléticas y una supresión del sistema inmune que ésta frecuentemente entre los Amish y los Finnish.[58][59][60] La variante mejor caracterizada es una transición A-G del nucleótido 70 que esta en una región bucle de dos bases 5' de un pseudoknot (estructura secundària del DNA) conservada. De todas maneras, muchas otras mutaciones entre ARNasa MRP también causan Cartilage-hair hypoplasia.
Enfermedad de Alzheimer
El antisense RNA BACE1-AS esta trascrito desde la banda opuesta del BACE1 y una regulación progresiva en los pacientes con Alzheimer.[61] BACE1-AS regula la expresión de BACE1 Mitjançant lo cual incrementa la estabilización del BACE1 mARN y genera BACE1 adicional a través de un mecanismo post-traduccional de prealimentación. Por el mismo mecanismo también se aumentan las concentraciones de beta-amieloide, el principal constituyente de las placas seniles. Las concentraciones BACE1-AS son elevadas en individuos con la enfermedad de Alzheimer y en ratones transgènicos con la proteína precursora de el amiloide.
miR-96 y Perdida de Audicion
Variaciones en la región de inicio del miR-96 maduro se ha asociado a una enfermedad autosòmica dominante de progresiva perdida de audición en humanos y ratones. En los ratones homocigotos mutantes eran profundamente sordos, no mostrando respuestas cocleares. En los humanos y en los ratones heterocigotos progresivamente pierden su habilidad de escuchar.[62] [63] [64]
Diferencia entre ARN funcional (fARN) y ncARN
Muchas publicaciones[65][66][67] han comenzado a utilizar el termino fARN, en comparación al ncARN, para describir regiones funcionales a nivel del ARN que podrían o no podrían mantener separados los ARN transcriptos. Por tanto, cada ncARN es un fARN, pero existen fARN (tales como riboswitches, elementos SEICS y otras regiones cis-reguladoras) que no son ncARN. sin embargo fARN también podría incluir mARN ya que este ARN codifica para proteínas y, por tanto, es funcional. A demás, ARNs evolucionados artificialmente también están englobados por el termino fARN. Algunas publicaciones[17] afirman que los términos ncARN y fARN son casi sinónimos.
Lease también
- Lista de RNAs
- Estructura de Acido Nucleico
- Rfam
- Riboswitch
- Ribozyme
- RNAs present in environmental samples
Referencias
- ↑ Hüttenhofer A, Schattner P, Polacek N (2005). «Non-coding RNAs: hope or hype?». Trends Genet 21 (5): pp. 289–97. doi:. PMID 15851066.
- ↑ Ban N, Nissen P, Hansen J, Moore P, Steitz T (2000). «The complete atomic structure of the large ribosomal subunit at 2.4 ångström resolution». Science 289 (5481): pp. 905–20. doi:. PMID 10937989.
- ↑ Reiner R, Ben-Asouli Y, Krilovetzky I, Jarrous N (2006). «A role for the catalytic ribonucleoprotein RNase P in RNA polymerase III transcription.». Genes Dev 20 (12): pp. 1621–35. doi:. PMID 16778078.
- ↑ Espinoza CA, Allen TA, Hieb AR, Kugel JF, Goodrich JA (2004). «B2 RNA binds directly to RNA polymerase II to repress transcript synthesis.». Nat Struct Mol Biol 11 (9): pp. 822–9. doi:. PMID 15300239.
- ↑ Hirota K, Miyoshi T, Kugou K, Hoffman CS, Shibata T, Ohta K (2008). «Stepwise chromatin remodelling by a cascade of transcription initiation of non-coding RNAs.». Nature 456 (7218): pp. 130–4. doi:. PMID 18820678.
- ↑ Saraiya AA, Wang CC (2008). «snoRNA, a novel precursor of microRNA in Giardia lamblia.». PLoS Pathog 4 (11): pp. e1000224. doi:. PMID 19043559.
- ↑ Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N, Meister G (2008). «A human snoRNA with microRNA-like functions.». Mol Cell 32 (4): pp. 519–28. doi:. PMID 19026782.
- ↑ Leygue E (2007). «Steroid receptor RNA activator (SRA1): unusual bifaceted gene products with suspected relevance to breast cancer.». Nucl Recept Signal 5: pp. e006. doi:. PMID 17710122.
- ↑ Zhang J, King ML (1996). «Xenopus VegT RNA is localized to the vegetal cortex during oogenesis and encodes a novel T-box transcription factor involved in mesodermal patterning.». Development 122 (12): pp. 4119–29. PMID 9012531.
- ↑ Kloc M, Wilk K, Vargas D, Shirato Y, Bilinski S, Etkin LD (2005). «Potential structural role of non-coding and coding RNAs in the organization of the cytoskeleton at the vegetal cortex of Xenopus oocytes.». Development 132 (15): pp. 3445–57. doi:. PMID 16000384.
- ↑ Jenny A, Hachet O, Závorszky P, Cyrklaff A, Weston MD, Johnston DS, Erdélyi M, Ephrussi A (2006). «A translation-independent role of oskar RNA in early Drosophila oogenesis.». Development 133 (15): pp. 2827–33. doi:. PMID 16835436.
- ↑ Gultyaev AP, Roussis A (2007). «Identification of conserved secondary structures and expansion segments in enod40 RNAs reveals new enod40 homologues in plants.». Nucleic Acids Res 35 (9): pp. 3144–52. doi:. PMID 17452360.
- ↑ Nakatani J, Tamada K, Hatanaka F, et al. (June 2009). «Abnormal behavior in a chromosome-engineered mouse model for human 15q11-13 duplication seen in autism». Cell 137 (7): pp. 1235–46. doi:. PMID 19563756.
Enlaces externos
- Comprehensive database of mammalian ncRNAs
- The Rfam Database — a curated list of hundreds of families of related ncRNAs.
- NONCODE.org — a free database of all kinds of noncoding RNAs (except tRNAs and rRNAs).
- Joint ncRNA Database — over 30,000 individual sequences from 99 species of bacteria, archaea and eukaryota
Plantilla:Nucleic acids
Categorías:- RNA
- Molecular genetics
Wikimedia foundation. 2010.