- T-box
-
T-box 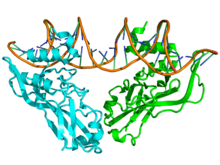
Estructura tridimensional de la proteína dimérica TBX3 (azul y verde) unida al ADN (marrón). Identificadores Símbolo T-box Pfam PF00907 InterPro IPR001699 PROSITE PS50252 SCOP 1xbr Estructuras en PDB: PDB 1h6f, PDB 1xbr T-box (en español "caja T") hace referencia a un grupo de factores de transcripción implicados en el desarrollo de las extremidades y del corazón.[1] En humanos y en algunos otros animales, defectos en la expresión génica del gen TBX5 pueden conducir a defectos en los pulgares y el septo ventricular, lo que da lugar a que no se produzca una correcta separación entre los ventrículos izquierdo y derecho del corazón.
Contenido
Organización del gen e historia evolutiva
El análisis de los genes de la caja T muestra que la mayoría de sus loci se encuentran distribuidos al azar en los genomas de los cordados, aunque hay algunos casos en los que están agrupados, por ejemplo TBX8 y TBX9 en Caenorhabditis elegans, TBX2 y TBX4 en cromosoma 11 y TBX3 y TBX5 en cromosoma 5 del ratón. La asociación entre los genes tardíos de las cajas T están conservados en otros mamíferos, como el hombre, en el caso de TBX2 y TBX4,y TBX3 y TBX5,que tienen una localización en los cromosomas 17 y 12 similar a la del ratón. El análisis filogenético de estos pares sugiere que provienen de la duplicación de un gen ancestral por entrecruzamiento (crossover) desigual entre dos alelos; en el caso de TBX2 y TBX4, y TBX3 y TBX5, este evento ocurrió hace por lo menos 600 millones de años.
Los genes de la caja T contienen múltiples exones, y la caja T generalmente está codificada por al menos cinco exones bastante separados. Por ejemplo, el gen humano TBX5 contiene ocho exones distribuidos en 53 kilobases del cromosoma 12. Como en otros genes, la unión entre el intrón y el exón está conservada, y en cambio la longitud del intrón varía entre diferentes especies. En la mayoría de los casos no se ha observado splicing alternativo, excepto en el caso de la proteína VegT/Antipodean de Xenopus, de la que se han hallado dos isoformas producidas por splicing alternativo del extremo 3'. Estas isoformas son específicas de tejido, una mesodérmica zigótica, y otra endodérmica embrionaria.
Función
Las proteínas de la caja T tienen un tamaño de 50 a 78 kDa, y se unen a la caja T, una región del ADN cuya secuencia consenso es 5'-TCACACCT-3'. Esta secuencia se puede repetir, y, de hecho, las repeticiones con dos o más secuencias consenso repetidas son las que tienen mayor afinidad por la proteína. Así mismo, las diversas repeticiones hacen a una secuencia T-box reconocible por una proteína T-box.
Las proteínas T-box tienen una función tanto represora como activadora. En todos los casos que se han estudiado se ha encontrado que la actividad reguladora se encuentra en el extremo C-terminal de la proteína. Sólo en el caso de Brachury y en su ortólogo en el pez cebra, se ha mapeado la regulación transcripcional. Curiosamente, el grado de conservación de estos ortólogos es bajo.
Los genes T-box tienen dos características que los hacen muy interesantes para estudiar los diferentes tipos celulares, sobre todo durante el desarrollo, y son necesarios para el correcto desarrollo de los tejidos. En los pocos casos en los que se ha estudiado la localización intracelular de las proteínas T-box, se han encontrado exclusivamente en el núcleo celular. Ese dato, junto con su actividad activadora/represora del ADN, indica que las proteínas T-box podrían ser importantes para la regulación del desarrollo. El hecho de que los alelos mutantes den un fenotipo mutante en heterocigosis (esto es, muestran haploinsuficiencia), indica que los niveles de proteína T-box son importantes para determinar su función. Adicionalmente, estudios de mutagénesis han demostrado que los genes T-box sólo tienen efecto en la célula que los expresa. Por ejemplo, Branchury se expresa en el mesodermo posterior y en la notocorda en desarrollo, y es imprescindible para la formación de esos tejidos en el ratón.
Los genes T-box también intervienen en el desarrollo tardío de tejidos específicos. Por ejemplo, TBX2, TBX3, TBX4 y TBX5 son cruciales en el desarrollo de las extremidades. TBX2 y TBX3 se expresan en los extremos anterior y posterior de las yemas de los miembros anteriores y posteriores. La expresión posterior de TBX3 es crucial para el desarrollo de otros elementos distales de las extremidades, como se observa en los pacientes a los que les falta TBX3, que padecen el síndrome ulnar-mamario y no tienen el hueso ulna ni dedos. A diferencia de la expresión simultánea de TBX2 y TBX3, sus homólogos TBX4 y TBX5, se expresan exclusivamente en las yemas posteriores y anteriores de las extremidades, respectivamente.
A pesar de que se desconocen las señales que dirigen la expresión diferencial de estos genes en los miembros anteriores y posteriores, parece que por lo menos intervendría la interpretación de las señales de los genes Hox, el llamado ``código Hox´´que se expresan en el mesodermo y que eventualmente da lugar a las células mesenquimáticas que migrarán a las yemas de las extremidades. Uno de esos genes es Ptx1. De todas maneras, la expresión es independiente de las señales que dirigen el crecimiento de la extremidad hacia fuera. Significativamente, estos genes no solo delimitan los territorios anterior y posterior de las extremidades, sino que también especifican el tipo de miembro anterior o posterior. Experimentos en los que se expresa incorrectamente TBX4 mediante retrovirus en la extremidad anterior de embriones de pollo, o TBX5 en la extremidad posterior, son capaces de transformar el tejido al tipo de miembro posterior y anterior, respectivamente. La transformación comprende tanto derivados esqueléticos del mesodermo, como el ectodermo que los recubre, y se desarrollan en grados que dependen de qué gen se esté expresando. TBX4 y TBX5, entonces, actúan como genes selectores en la yema de la extremidad, definiendo qué tipo de extremidad se debe desarrollar. Finalmente, las mutaciones en TBX5 humano afectan al crecimiento de las extremidades anteriores y al desarrollo del corazón. Curiosamente, las mutaciones sin sentido en TBX5 producen anomalías primarias en las extremidades, mientras que otras anomalías del síndrome de Holt-Oram como el desarrollo aberrante del corazón, se producen predominantemente como resultado de mutaciones de cambio de sentido, que altera un aminoácido que contacta con el surco mayor del ADN. Esto sugiere que genes diana específicos de tejido son afectados por mutaciones en distintos residuos del gen T-box que codifica TBX5.
Se han relacionado otras mutaciones en proteínas T-box con el síndrome de DiGeorge, una enfermedad compleja que implica anomalías en las arterias coronarias. También se ha descrito que TBX2 está amplificado en determinados tipos de cáncer de mama.
Proteínas T-box
A continuación se indican las proteínas T-box descritas, así como los genes que las codifican:
- TBR1 TBR1)
- TBX1 (TBX1)
- TBX2 (TBX2)
- TBX3 (TBX3)
- TBX4 (TBX4)
- TBX5 (TBX5)
- TBX6 (TBX6)
- TBX10 (TBX10)
- TBX15 (TBX15)
- TBX18 (TBX18)
- TBX19 (TBX19)
- TBX20 (TBX20)
- TBX21 (TBX21)
- TBX22 (TBX22)
Retos para el futuro
A pesar de que el papel de los genes concretos T-box es muy importante, se sabe muy poco sobre las rutas bioquímicas en las que están implicados. Entonces, un punto importante en futuras investigaciones debe ser identificar los factores que modulan la transcripción de estos genes corriente arriba y corriente abajo.
A nivel celular, las mutaciones han demostrado que estas proteínas se necesitan en fenómenos del desarrollo variados, pero la función exacta de los genes T-box, incluidas cuestiones de redundancia genética, aún está por descubrir. Por ejemplo, en el síndrome ulnar-mamario, no se sabe por qué no hay defectos en las rodillas, cuando TBX normalmente se expresa en esa zona. Esto se puede deber a una redundancia con otros genes T-box que también se expresan en esa región, como TBX2 o TBX4.
Con el fin de conocer en profundidad la función de genes T-box individuales, también será necesario generar series alélicas, que serían útiles para el estudio del síndrome de Holt-Oram, y mutaciones condicionales.
Finalmente, se conoce relativamente poco acerca del procesamiento post-transcripcional, el recambio, y la estabilidad de cualquier proteína T-box. Adicionalmente, solo en los casos de TBX5, TBR1, y MGA, se conocen dominios de interacción proteína-proteína y, con la excepción de MGA, no se sabe el significado biológico de esas interacciones.
Véase también
- Homeobox
- Genes de desarrollo en plantas: G-box, I-box, W-box, Z-box
Referencias
- ↑ Wilson V, Conlon FL (2002). «The T-box family». Genome Biol. 3 (6): pp. REVIEWS3008. doi:. PMID 12093383.
Enlaces externos
Wikimedia foundation. 2010.
