- Complejo exosoma
-
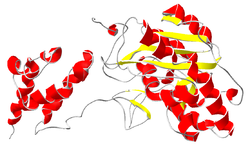
 Estructura de cintas del complejo del exosoma humano. Véase la leyenda más abajo.
Estructura de cintas del complejo del exosoma humano. Véase la leyenda más abajo.
El complejo exosoma, también denominado complejo PM/Scl o solamente exosoma, es un complejo multiproteico capaz de degradar diversos tipos de ARNs. Los complejos de exosoma pueden encontrarse en células eucariotas y archaeas, mientras que en las bacterias es un complejo más simple, el degradasoma, el que lleva a cabo funciones similares.
El núcleo del complejo tiene una estructura anular formada por seis miembros, al que se acoplan otras proteínas. En las células eucariotas está presente en el citoplasma y en el núcleo celular, especialmente en el nucléolo, aunque las proteínas que interaccionan con el complejo en estos compartimentos son diferentes, con el fin de regular su actividad de degradación del ARN de arreglo a la diferente naturaleza de los sustratos presentes.
Estos sustratos comprenden ARN mensajero, ARN ribosómico y muchas especies de ARN de pequeño tamaño. El exosoma tiene una función exorribonucleolítica, lo que significa que degrada el ARN comenzando por uno de los extremos (el llamado extremo 3' en este caso) en lugar de escindir el ARN en lugares específicos.
Aunque no existe relación causal entre el complejo y ninguna de las enfermedades conocidas, algunas de sus proteínas son el antígeno de autoanticuerpos responsables de algunas enfermedades autoinmunes (especialmente del síndrome de solapamiento PM/Scl) y de algunas quimioterapias antimetabolíticas que bloquean la actividad del complejo.
Contenido
Descubrimiento
El exosoma fue descubierto como una ribonucleasa en 1997 en la levadura Saccharomyces cerevisiae, que es empleada en muchos laboratorios como organismo modelo.[1] No mucho después, en 1999, se comprobó que el exosoma era de hecho el equivalente del complejo ya descrito en células humanas, conocido como complejo PM/Scl, que había sido identificado como un autoantígeno en pacientes con ciertas enfermedades autoinmunes en años anteriores.[2] La purificación del complejo humano permitió la identificación de más proteínas del exosoma y eventualmente la caracterización de todos sus componentes.[3] [4] En 2001 la creciente cantidad de datos procedentes de los distintos proyectos genoma que comenzaron a estar disponibles, permitieron la predicción de proteínas del exosoma de archaeas, aunque tuvieron que transcurrir otros dos años antes de que se purificara el primer exosoma de uno de estos organismos.[5] [6]
Estructura
Proteínas centrales
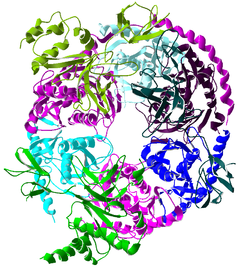
 Vista desde arriba (imagen superior) y desde un lateral (imagen inferior) de la estructura cristalina del complejo de exosoma humano. Ver leyenda más abajo.
Vista desde arriba (imagen superior) y desde un lateral (imagen inferior) de la estructura cristalina del complejo de exosoma humano. Ver leyenda más abajo.
El núcleo del complejo tiene una estructura anular que comprende seis proteínas, todas ellas pertenecientes al mismo tipo de ribonucleasa, las proteínas similares a las ARNasas PH.[7] En las archaea hay dos proteínas distintas de este tipo (llamadas Rrp41 y Rrp42), cada una de ellas presente tres veces en orden alterno. Los complejos de exosoma eucariota tienen seis proteínas diferentes que forman la estructura anular.[8] [9] De esas seis proteínas eucariotas, tres son semejantes a la proteína arqueota Rrp41 y las otras tres guardan mayor similitud con Rrp42.[10]
Situadas en la parte superior del anillo se disponen tres proteínas que tienen un dominio de unión a ARN de tipo S1 (RBD). Dos proteínas, además, tienen un domino de homología K (dominio KH).[7] En eucariotas se unen tres proteínas S1 diferentes al anillo, mientras que en achaea pueden formar parte del exosoma una o dos tipos de éstas (aunque siempre hay tres subunidades S1 unidas al complejo).[11]
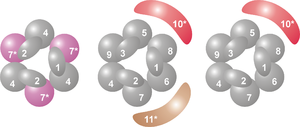
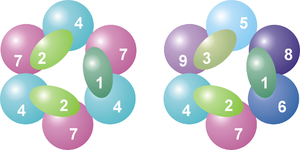 Subunidades y organización de un complejo de exosoma arqueota (izquierda) y eucariota (derecha). Se numeran las distintas proteínas, mostrando que el exosoma arqueota contiene cuatro proteínas distintas, mientras que en el caso eucariota son nueve. Ver la leyenda más abajo.
Subunidades y organización de un complejo de exosoma arqueota (izquierda) y eucariota (derecha). Se numeran las distintas proteínas, mostrando que el exosoma arqueota contiene cuatro proteínas distintas, mientras que en el caso eucariota son nueve. Ver la leyenda más abajo.
La estructura en anillo es muy similar a la de las ARNasas PH y a la de las polinucleótido fosforilasas (PNPasa). En las bacterias, la ARNasa PH que está implicada en el procesamiento del ARNt forma un anillo examérico consistente en seis ARNasas PH idénticas.[12] En el caso de la PNPasa, que es una proteína fosforolítica que degrada ARN y que se encuentra tanto en bacterias como en cloroplastos y mitocondrias de algunos organismos eucariotas, serían parte de la misma proteína dos dominios ARNasa PH y también un dominio S1 y otro de tipo KH de unión a ARN. Esta proteína forma un complejo trimérico que adopta una estructura casi idéntica a la del exosoma.[13] Debido a la gran similitud entre ambos dominios de proteína y también de su estructura, se piensa que estos complejos están relacionados evolutivamente y tienen un antepasado común.[14] En las bacterias existe una ARNasa PH diferente implicada en el procesamiento del ARN de transferencia, del que se ha visto que adopta una estructura anular similar de seis unidades, pero que en este caso las unidades son idénticas.[15] Las proteínas similares a la ARNasa PH, las propias ARNasas PH y las PNPasas pertenecen todas ellas a la familia de las ARNasas y las exorribonucleasas fosforolíticas, lo cual significa que utilizan fosfato inorgánico para eliminar nucleótidos del extremo 3' de las moléculas de ARN.[7]
Proteínas asociadas
Además de las nueve proteínas nucleares, otras dos proteínas se ven frecuentemente asociadas con el complejo de exosoma en los organismos eucariotas. Una de ellas es Rrp44, una RNasa hidrolítica, que pertenece a la familia ARNasa R de exorribonucleasas hidrolíticas (nucleasas que utilizan el agua para escindir nucleótidos). En levaduras, la Rrp44 está asociada con todos los complejos de exosoma y tiene un papel crucial en la actividad de estos complejos.[16] Cabe destacar que, aunque existe una proteína homóloga humana, no se ha encontrado ninguna evidencia de que ésta se encuentre asociada al complejo de exosoma humano.[7]
La segunda proteína común asociada se llama Rrp6 (en levaduras) o PM/Scl-100 (en humanos). Como la Rrp44, esta proteína también es una exorribonucleasa hidrolítica, pero en este caso pertenece a la familia de proteínas ARNasa D.[17] La proteína PM/Scl-100 compone la subunidad más común de los complejos de exosoma en el núcleo de las células, pero también forma parte del complejo de exosoma citoplasmático.[18]
Proteínas reguladoras
Además de estas dos subunidades estrechamente unidas, muchas proteínas interactúan con el complejo exosómico tanto en el citoplasma como en el núcleo. Estas proteínas que se unen de forma laxa podrían regular la actividad y la especificidad del complejo de exosoma. En el citoplasma, el exosoma interactúa con proteínas que se unen a elementos ricos en AU (ARE, p.ej. KRSP y TTP), que pueden promover o impedir la degradación de los ARNm. El exosoma nuclear se asocia con las proteínas que unen ARN (p.ej. MPP6 en humanos y Rrp47/C1D en humanos y levaduras) que controlan el procesamiento del ARN ribosómico.[7]
Además de proteínas individuales, también interactúan con el exosoma otros complejos proteicos. Uno de ellos el el complejo Ski, que incluye una ARN helicasa (Ski2) y está implicado en la degradación del ARNm.[19] En el núcleo, el procesamiento del ARNr y snoARN por el exosoma está mediado por el complejo TRAMP, que posee tanto la helicasa de ARN Mtr4 como actividad de poliadenilación (Trf4).[20]
Función
Función enzimática
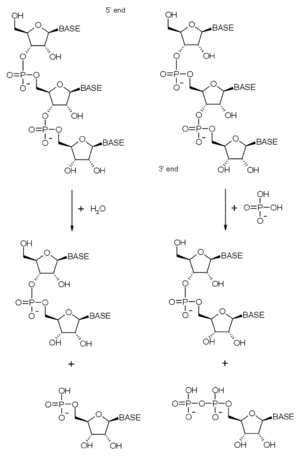 Diagramas de reacción de la degradación hidrolítica (izquierda) y fosforolítica (derecha) del extremo 3' del ARN.
Diagramas de reacción de la degradación hidrolítica (izquierda) y fosforolítica (derecha) del extremo 3' del ARN.
Como se afirmó anteriormente, el complejo exosómico contiene muchas proteínas con dominios ribonucleasa. Estos tienen una actividad exorribonulcleasa 3'→ 5', lo que significa que las enzimas degradan las moléculas de ARN a partir de su extremo 3'. El complejo contiene exorribonucleasas y en eucariotas también se presentan exorribonucleasas hidrolíticas (las proteínas con dominios ARNasa D y R). Las enzimas fosforolíticas usan fosfato inorgánico para escindir los enlaces fosfodiéster, liberando nucleótidos monofosfato.
En arqueotas, la subunidad Rrp41 del complejo es la exorribonucleasa fosforolítica. El anillo de esta enzima presenta tres copias de esta proteína, que son las responsables de la actividad del complejo.[9] En eucariotas, ninguna de las subunidades ARNasa PH ha mantenido esta actividad catalítica, lo cual supone que la estructura anular central del exosoma humano no tiene ninguna proteína enzimáticamente activa.[21] En levaduras esto se compensa por una de las enzimas hidrolíticas asociadas, la Rrp44, que es la responsable de la mayor parte de la actividad ribonucleasa del exososoma,[22] pero esta subunidad hidrolítica en particular puede estar restringida a las levaduras, puesto que no se ha encontrado ninguna proteína homóloga a Rrp44 en el exosoma humano (y tampoco en arqueotas).[23] [24]
Se puede asociar todavía otra enzima hidrolítica en humanos y levaduras con el complejo (Rrp6), que contribuye a la actividad del exosoma de levaduras y es la única responsable de la actividad del complejo humano. Aunque originalmente se pensaba que las proteínas con dominio S1 tenían también actividad hidrolítica 3'→5', trabajos posteriores lo han puesto en duda, afirmando que estas proteínas podrían desempeñar tan solo un papel en la unión de sustratos antes de su degradación por el complejo.[22]
Sustratos
El exosoma está implicado en la degradación y procesamiento de una gran variedad de especies de ARN. En el citoplasma celular está implicado en el reemplazamiento (turn-over) de las moléculas de ARN mensajero. El complejo puede degradar moléculas de ARNm que han sido marcadas para su degradación porque contienen errores, mediante interacciones con proteínas de la ruta mediada por mutación terminadora o por la ruta mediada por mutación "non-stop". Por otra parte, los ARNm se degradan como parte de su ciclo normal. Existen varias proteínas que estabilizan o desestabilizan moléculas de ARNm uniéndose a los elementos ricos en AU de los extremos 3' no traducidos (3'UTR) y que interactúan con el complejo del exosoma.[25] [26] [27]
En el núcleo celular, el exosoma se requiere para el correcto procesamiento de varios snARN's.[28] Finalmente, el nucléolo es el compartimento donde se encuentran la mayor parte de los complejos de exosoma, donde desempeñan un papel en el procesamiento del ARN ribosómico 5,8S (la primera función del exosoma identificada) y de varios snoARN's.[1] [29] [28]
Aunque la mayor parte de las células tienen otras enzimas con capacidad de degradar ARN tanto por su extremo 3' como por su extremo 5', el exosoma es esencial para la supervivencia celular. Cuando se detiene o reduce la expresión de las proteínas del exosoma por medios artificiales, por ejemplo, mediante ARN interferente, el crecimiento se detiene y las células finalmente mueren. Tanto las proteínas centrales del complejo como las dos proteínas principales asociadas son esenciales.[30]
Las bacterias no tienen exosoma, aunque sus funciones son asumidas por un complejo similar, llamado degradasoma del que forma parte la proteína polinucleótido fosforilasa.[31]
Patología
Autoinmunidad
El complejo exosómico es el antígeno de los autoanticuerpos responsables de diversas enfermedades autoinmunes. Estos autoanticuerpos se encuentran principalmente en personas que sufren el síndrome de solapamiento PM-Scl, una enfermedad autoinmune en que los pacientes sufren una esclerodermia de base acompañada o bien de polimiositis o bien de dermatomiositis.[32] Los autoanticuerpos se pueden detectar en el suero de los pacientes mediante diversos ensayos. En el pasado, el método más común fue el de inmunodifusión doble de Ouchterlony utilizando extractos de timo de ternera, inmunofluorescencia en células HEp-2 o inmunoprecipitación a partir de extractos celulares humanos. En los ensayos de inmunoprecipitación con sueros anti-exosoma se precipita un conjunto de proteínas característico. En los años anteriores a la caracterización del exosoma, se denominó a este precipitado complejo PM-Scl.[33] Los sueros de estos pacientes suelen mostrar una característica tinción del nucléolo celular, lo que alimentó la hipótesis de que el antígeno reconocido por los autoanticuerpos era importante en la síntesis ribosomal.[34] Más recientemente se ha conseguido disponer de proteínas exosómicas que han podido ser utilizadas para desarrollar inmunoanálisis en línea (LIA) y ensayos de ELISAs para detectar estos anticuerpos.[7]
En estas enfermedades, los anticuerpos se dirigen principalmente contra dos de las proteínas del complejo, denominadas PM/Scl-100 (la proteína similar a una ARNasa D) y PM/Scl-75 (una de las proteínas similares a las ARNasas PH del anillo), encontrándose anticuerpos contra estas proteínas en aproximadamente un 30% de los pacientes que sufren del síndrome.[35] Aunque estas dos proteínas son el ligando principal de los autoanticuerpos, también pueden serlo otras subunidades o proteínas asociadas, como C1D.[36] [37] Actualmente, el método con mayor sensibilidad para detectar estos autoanticuerpos consiste en el uso de un péptido derivado de la proteína PM/Scl-100 como antígeno en una ensayo ELISA, en lugar de utilizar proteínas completas. Mediante este método, los autoanticuerpos se encuentran en más del 55% de los pacientes con el síndrome de solapamiento, aunque también pueden ser detectados en pacientes que sufren tanto de esclerodermia, como de polimiositis o de dermatomiositis por separado.[38]
Puesto que los autoanticuerpos se encuentran principalmente en pacientes que tienen características de varias enfermedades autoinmunes distintas, los síntomas clínicos de estos pacientes pueden variar a lo largo de un rango bastante amplio. Los síntomas que se ven con más frecuencia son los típicos de estas enfermedades, entre ellos el signo de Raynaud, artritis, miositis y esclerodermia.[39] El tratamiento de estos pacientes es sintomático y similar al tratamiento de cada enfermedad autoinmune por separado, lo que supone la administración de fármacos inmunosupresores o inmunomoduladores.[40]
Cáncer
Se ha demostrado que el exosoma es inhibido por el antimetabolito 5-fluorouracilo, utilizado también en el tratamiento quimioterapéutico del cáncer. Este es uno de los tratamientos más eficaces contra los tumores sólidos. Cuando este fármaco es administrado a levaduras, se pueden observar defectos en el procesamiento del ARN ribosómico idénticos a los que se aprecian cuando se bloquea dicho procesamiento mediante diversas estrategias utilizadas en biología molecular. La ausencia de un correcto procesado de los ribosomas es letal para estas células, lo que explica su efecto antimetabólico.[41]
Lista de subunidades
Leyenda Nombre general Dominios Homo sapiens S. cerevisiae Archaea Masa molecular (kDa) Gen humano Gen levadura 1 Csl4 S1 RBD hCsl4 Csl4p/Ski4p Csl4 21-32 EXOSC1 YNL232W 2 Rrp4 S1/KH RBD hRrp4 Rrp4p Rrp4 28-39 EXOSC2 YHR069C 3 Rrp40 S1/KH RBD hRrp40 Rrp40p (Rrp4)[42] 27-32 EXOSC3 YOL142W 4 Rrp41 ARNasa PH hRrp41 Rrp41p/Ski6p Rrp41 26-28 EXOSC4 YGR195W 5 Rrp46 ARNasa PH hRrp46 Rrp46p (Rrp41)[42] 25-28 EXOSC5 YGR095C 6 Mtr3 ARNasa PH PH hMtr3 Mtr3p (Rrp41)[42] 24-37 EXOSC6 YGR158C 7 Rrp42 ARNasa PH hRrp42 Rrp42p Rrp42 29-32 EXOSC7 YDL111C 8 Rrp43 ARNasa PH OIP2 Rrp43p (Rrp42)[42] 30-44 EXOSC8 YCR035C 9 Rrp45 ARNasa PH PM/Scl-75 Rrp45p (Rrp42)[42] 34-49 EXOSC9 YDR280W 10 Rrp6 ARNasa D PM/Scl-100 Rrp6p n/a 84-100 EXOSC10 YOR001W 11 Rrp44 ARNasa R (hDis3)[43] Rrp44p/Dis3p n/a 105-113 KIAA1008 YOL021C - 42: En arqueotas varias proteínas del exosoma están presentes en múltiples copias para formar el núcleo completo del complejo exosómico.
- 43: Aunque la proteína Rrp44 es parte del complejo en levadura, su homólogo humano, hDis3, no se ha encontrado nunca asociado con el complejo humano.
Véase también
- Proteasoma, el principal complejo celular de degradación de proteínas.
- Espliceosoma, un complejo implicado en el splicing de ARN, que también posee una estructura anular de unión al ARN.
Referencias
- ↑ a b Mitchell et al. (1997). «The Exosome: A Conserved Eukaryotic RNA Processing Complex Containing Multiple 3′→5′ Exoribonucleases». Cell 91 (4): pp. 457–466. doi:. PMID 9390555.
- ↑ Allmang et al. (1999). «The yeast exosome and human PM-Scl are related complexes of 3'→ 5' exonucleases». Genes and Development 13 (16): pp. 2148–58. doi:. PMID 10465791.
- ↑ Brouwer et al. (2001). «Three novel components of the human exosome». Journal of Biological Chemistry 276: pp. 6177–84. doi:. PMID 11110791.
- ↑ Chen et al. (2001). «AU binding proteins recruit the exosome to degrade ARE-containing mRNAs». Cell 107: pp. 451–64. doi:. PMID 11719186.
- ↑ Koonin et al. (2001). «Prediction of the archaeal exosome and its connections with the proteasome and the translation and transcription machineries by a comparative-genomic approach». Genome Research 11 (2): pp. 240–52. doi:. PMID 11157787.
- ↑ Evguenieva-Hackenberg et al. (2003). «An exosome-like complex in Sulfolobus solfataricus». EMBO Reports 4 (9): pp. 889–93. doi:. PMID 12947419.
- ↑ a b c d e f Schilders et al. (2006). «Cell and molecular biology of the exosome: how to make or break an RNA». International review of cytology 251: pp. 159–208. doi:. PMID 16939780.
- ↑ Lorentzen et al. (2005). «The archaeal exosome core is a hexameric ring structure with three catalytic subunits». Nature Structural & Molecular Biology 12: pp. 575–81. doi:. PMID 15951817.
- ↑ a b Shen et al. (2006). «A view to a kill: structure of the RNA exosome». Cell 127: pp. 1093–5. doi:. PMID 17174886.
- ↑ Raijmakers et al. (2002). «Protein-protein interactions between human exosome components support the assembly of RNase PH-type subunits into a six-membered PNPase-like ring». Journal of Molecular Biology 323: pp. 653–63. doi:. PMID 12419256.
- ↑ Walter et al. (2006). «Characterization of native and reconstituted exosome complexes from the hyperthermophilic archaeon Sulfolobus solfataricus». Molecular Microbiology 62: pp. 1076–89. doi:. PMID 17078816.
- ↑ Ishii et al. (2003). «Crystal structure of the tRNA processing enzyme RNase PH from Aquifex aeolicus». Journal of Biological Chemistry 278: pp. 32397–404. doi:. PMID 12746447.
- ↑ Symmons et al. (2000). «A duplicated fold is the structural basis for polynucleotide phosphorylase catalytic activity, processivity, and regulation». Structure 8: pp. 1215–26. doi:. PMID 11080643.
- ↑ Lin-Chao et al. (2007). «The PNPase, exosome and RNA helicases as the building components of evolutionarily-conserved RNA degradation machines». Journal of Biomedical Science 14: pp. 523–32. doi:.
- ↑ Harlow et al. (2004). «Crystal structure of the phosphorolytic exoribonuclease RNase PH from Bacillus subtilis and implications for its quaternary structure and tRNA binding». Protein Science 13: pp. 668–77. doi:. PMID 14767080.
- ↑ Schneider et al. (2007). «The exosome subunit Rrp44 plays a direct role in RNA substrate recognition». Molecular Cell 27: pp. 324–31. doi:. PMID 17643380.
- ↑ Mian et al. (1997). «Comparative sequence analysis of ribonucleases HII, III, II PH and D». Nucleic Acids Research 25: pp. 3187–3195. doi:. PMID 9241229.
- ↑ Raijmakers et al. (2004). «The exosome, a molecular machine for controlled RNA degradation in both nucleus and cytoplasm». European Journal of Cell Biology 83: pp. 175–83. doi:. PMID 15346807.
- ↑ Wang et al. (2005). «Domain interactions within the Ski2/3/8 complex and between the Ski complex and Ski7p». RNA 11: pp. 1291–302. doi:. PMID 16043509.
- ↑ LaCava et al. (2005). «RNA degradation by the exosome is promoted by a nuclear polyadenylation complex». Cell 121: pp. 713–24. doi:. PMID 15935758.
- ↑ Liu et al. (2007). «Erratum: Reconstitution, activities, and structure of the eukaryotic RNA exosome». Cell 131: pp. 188–189. doi:. PMID 17174896.
- ↑ a b Dziembowski et al. (2007). «A single subunit, Dis3, is essentially responsible for yeast exosome core activity». Nature Structural & Molecular Biology 14: pp. 15–22. doi:. PMID 17173052.
- ↑ Liu et al. (2006). «Reconstitution, activities, and structure of the eukaryotic RNA exosome». Cell 127: pp. 1223–37. doi:. PMID 1717489.
- ↑ Lorentzen et al. (2005). «Structural basis of 3' end RNA recognition and exoribonucleolytic cleavage by an exosome RNase PH core». Molecular Cell 20: pp. 473–81. doi:. PMID 16285928.
- ↑ LeJeune et al. (2003). «Nonsense-mediated mRNA decay in mammalian cells involves decapping, deadenylating, and exonucleolytic activities». Molecular Cell 12: pp. 675–87. doi:. PMID 14527413.
- ↑ Wilson et al. (2007). «A genomic screen in yeast reveals novel aspects of nonstop mRNA metabolism». Genetics 177: pp. 773. doi:. PMID 17660569.
- ↑ Lin et al. (2007). «Localization of AU-rich element-containing mRNA in cytoplasmic granules containing exosome subunits». Journal of Biological Chemistry 282: pp. 19958–68. doi:. PMID 17470429.
- ↑ a b Allmang et al. (1999). «Functions of the exosome in rRNA, snoRNA and snRNA synthesis». EMBO Journal 18: pp. 5399–410. doi:. PMID 10508172.
- ↑ Schilders et al. (2005). «MPP6 is an exosome-associated RNA-binding protein involved in 5.8S rRNA maturation». Nucleic Acids Research 33: pp. 6795–804. doi:. PMID 16396833.
- ↑ van Dijk et al. (2007). «Human cell growth requires a functional cytoplasmic exosome, which is involved in various mRNA decay pathways». RNA 13: pp. 1027–35. doi:. PMID 17545563.
- ↑ Carpousis AJ (2002). «The Escherichia coli RNA degradosome: structure, function and relationship in other ribonucleolytic multienzyme complexes». Biochem. Soc. Trans. 30 (2): pp. 150–5. doi:. PMID 12035760. http://www.biochemsoctrans.org/bst/030/0150/bst0300150.htm.
- ↑ J.E. Pope (2002). «Scleroderma overlap syndromes». Current Opinion in Rheumatology 14: pp. 704–10. doi:. PMID 12410095.
- ↑ Gelpi et al. (1991). «Identification of protein components reactive with anti-PM/Scl autoantibodies». Clinical and Experimental Immunology 81: pp. 59–64. PMID 2199097.
- ↑ Targoff et al. (1985). «Nucleolar localization of the PM-Scl antigen». Arthritis & Rheumatism 28: pp. 226–30. doi:. PMID 3918546.
- ↑ Raijmakers et al. (2004). «PM-Scl-75 is the main autoantigen in patients with the polymyositis/scleroderma overlap syndrome». Arthritis & Rheumatism 50: pp. 565–9. doi:. PMID 14872500.
- ↑ Brouwer et al. (2002). «Autoantibodies directed to novel components of the PM/Scl complex, the human exosome.». Arthritis Research 4: pp. 134–8. doi:. PMID 11879549.
- ↑ Schilders et al. (2007). «C1D is a major autoantibody target in patients with the polymyositis-scleroderma overlap syndrome». Arthritis & Rheumatism 56: pp. 2449–54. doi:. PMID 17599775.
- ↑ Mahler et al. (2005). «Clinical evaluation of autoantibodies to a novel PM/Scl peptide antigen». Arthritis Research & Therapy 7: pp. R704–13. doi:. PMID 15899056.
- ↑ Mahler et al. (2007). «Novel aspects of autoantibodies to the PM/Scl complex: Clinical, genetic and diagnostic insights». Autoimmunity Reviews 6: pp. 432–7. doi:. PMID 17643929.
- ↑ Jablonska et al. (1998). «Scleromyositis: a scleroderma/polymyositis overlap syndrome». Clinical Rheumatology 17: pp. 465–7. doi:. PMID 9890673.
- ↑ Lum et al. (2004). «Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes». Cell 116: pp. 121–37. doi:. PMID 14718172.
- ↑ a b c d e En arqueotas varias proteínas del exosoma están presentes en múltiples copias para formar el núcleo completo del complejo exosómico.
- ↑ Aunque la proteína Rrp44 es parte del complejo en levadura, su homólogo humano, hDis3, no se ha encontrado nunca asociado con el complejo humano.
Bibliografía
- Vanacova y col. (2007). «The exosome and RNA quality control in the nucleus». EMBO reports 8: pp. 651–657. doi:. http://npg.nature.com/embor/journal/v8/n7/full/7401005.html.
- Houseley y col. (2006). «RNA-quality control by the exosome». Nature Reviews Molecular Cell Biology 7: pp. 529–539. doi:. http://www.nature.com/nrm/journal/v7/n7/abs/nrm1964.html. –-- subscription required
- Büttner y col. (2006). «The exosome: a macromolecular cage for controlled RNA degradation». Molecular Microbiology 61: pp. 1372–1379. doi:. http://www.blackwell-synergy.com/doi/abs/10.1111/j.1365-2958.2006.05331.x.
- Lorentzen y col. (2006). «The Exosome and the Proteasome: Nano-Compartments for Degradation». Cell 125: pp. 651–654. doi:.
- G.J.M. Pruijn (2005). «Doughnuts dealing with RNA». Nature Structural & Molecular Biology 12: pp. 562–564. doi:. http://www.nature.com/nsmb/journal/v12/n7/full/nsmb0705-562.html.
Enlaces externos
Categorías:- ARN
- Complejos proteicos
Wikimedia foundation. 2010.