- Chip de ADN
-
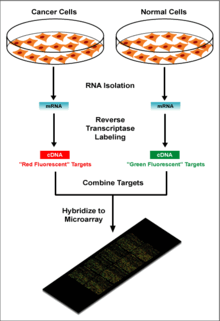
Un chip de ADN (del inglés DNA microarray) es una superficie sólida a la cual se une una colección de fragmentos de ADN. Las superficies empleadas para fijar el ADN son muy variables y pueden ser de vidrio, plástico e incluso de silicio. Los chips de ADN se usan para analizar la expresión diferencial de genes, monitorizándose los niveles de miles de ellos de forma simultánea. Su funcionamiento consiste, básicamente, en medir el nivel de hibridación entre la sonda específica (probe, en inglés), y la molécula diana (target), indicándose generalmente mediante fluorescencia y analizándose por análisis de imagen, lo cual nos indicará el nivel de expresión del gen.
Suelen utilizarse para identificar genes con una expresión diferencial bajo condiciones distintas. Por ejemplo, para detectar genes que producen ciertas enfermedades mediante la comparación de los niveles de expresión entre células sanas y células que están desarrollando ciertos tipos de enfermedades.
Contenido
Antecedentes históricos
La tecnología del chip de ADN tiene su origen en una técnica muy usada en biología molecular, el Southern blot. En la era pre-genómica la biología estudiaba los genes individualmente, uno a uno, por lo que los podía estudiar a fondo. Lo que caracteriza la era post-genómica no es lo que se puede medir sino la cantidad de mediciones simultáneas que se pueden realizar. Para cumplir este objetivo y poder estudiar muchos genes a la vez fue necesario un cambio de paradigma : con los mismos recursos, obtener una imagen de menor resolución pero con una perspectiva más general.
Fabricación
Los chips de ADN se fabrican usando una gran variedad de tecnologías. El gran desarrollo de esta técnica ha llegado al normalizarse el uso de robots que se encargan de la mayor parte del proceso de manipulación de los chips (sintetizar el ADN molde que se une al chip, unirlo, añadir los reactivos necesarios, etc.).
Los chips de ADN se pueden usar para detectar ARN, que puede o no ser traducido a proteínas. Los científicos se refieren a esta clase de análisis como "análisis de expresión" o "análisis del transcriptoma". Se suele retrotranscribir el ARN a ADN para estos casos, de manera que lo que usamos como muestra es ADNc.
El uso de chips de ADN para estudiar la expresión de diversos genes fue publicado en 1995,[1] en la prestigiosa revista científica Science y el primer organismo eucariota con todo el genoma (Saccharomyces cerevisiae) dispuesto en un chip de ADN fue publicado en 1997[2] en la misma revista.
Tipos
Microarrays de dos canales
En este tipo de chips de ADN (en inglés Spotted microarrays) las pruebas son oligonucleótidos, ADN complementario (ADNc) o pequeños fragmentos de Reacción en cadena de la polimerasa, que corresponden con ARN mensajero (ARNm). En este tipo de chip de ADN se híbrida el ADNc de dos condiciones que son marcados, cada uno de esas condiciones con dos fluoróforos diferentes. Las condiciones son mezcladas e hibridades en el mismo chip de ADN. Una vez realizado este primer paso se procede al escaneo del resultado y a la visualización del mismo. De esta forma se pueden observar genes que se activan o se reprimen en distintas condiciones. La contrapartida de estos experimentos es que no se pueden observar niveles absolutos en la expresión y requieren qPCR para analysis quantitativo absoluto.
Chips de ADN de oligonucleótidos En los chips de ADN de oligonuleótidos o micromarreglos de canal único, las pruebas son designadas a partes de una secuencia conocida o un ARNm predicho. Estos chips de ADNs dan estimaciones del nivel de expresión, pero distintas condiciones no pueden ser observadas en una misma matriz, por lo que por cada condición se ha de utilizar un chip.
Idea principal : las sondas son sintetizadas "in situ" (en el chip).
No están basadas en la hibridación competitiva. Ésto quiere decir : un chip, una muestra. Las secuencias son construidas en la superficie del chip mediante el elongamiento secuencial de una cadena en crecimiento con un sólo nucleótido utilizando fotolitografía.
GeneChips de Affymetrix
•Affymetrix es la compañía lider en este tipo de chips. •Se denominan genéricamente "GeneChips". •Cada gen representado por un conjunto de secuencias cortas que lo caracterizan. •Algunos chips: genomas completos con más de 50.000 grupos de sondas.
Chips de ADN para Genotipado
Los chips de ADN pueden ser utilizadas para "leer" las secuencias de un genoma particular en determinadas posiciones.
Los SNP arrays son un tipo particular de matrices que son usadas para identificar variaciones individuales y a través de poblaciones. Los oligonucleotidos pequeños son capaces de identificar polimorfismos de un sólo nucleótido (en inglés SNPs, single nucleotide polymorphisms) que podrían ser los responsables de variaciones genéticas dentro de una población, la fuente de susceptibilidad a distintas enfermedades genéticas e incluso a ciertos tipos de cáncer. En general, la aplicación de estas técnicas de genotipado es forense, ya que son rápidas en descubrir o medir la predisposición de enfermedades o incluso permitir el uso de ciertos medicamentos para tratar ciertas enfermedades según sea el ADN del enfermo o donante. Los chips de ADN de SNPs son también utilizadas para identificación de mutaciones somáticas en cáncer, sobre todo la pérdida de heterocigosis, la amplificación o la deleción de regiones de ADN en el genoma individual de pacientes afectados, es decir la detección de aberraciones cromosómicas..
Aplicaciones
Los chips de ADN se han aplicado al estudio de casi cualquier tipo de problema biológico. El numero de publicaciones anuales es muy alto y continúa creciendo. Algunas de sus aplicaciones más frecuentes son :
- Estudio de genes que se expresan diferencialmente entre varias condiciones (sanos/enfermos, mutantes/salvajes, tratados/no tratados).
- Clasificación molecular en enfermedades complejas identificación de genes característicos de una patología (firma o “signature”).
- Predicción de respuesta a un tratamiento.
- Detección de mutaciones y polimorfismos de un único gen (SNP).
Bases de datos públicas para el análisis de chips de ADN
- Stanford Microarray database
- Yale Microarray Database
- UNC Microarray database
- MUSC database
- Gene Expression Omnibus - NCBI
- ArrayExpress - EBI
- University of Tennessee Microarray Database
- microarray databases Repositorio de bases de datos de microarrays
Lista de compañías que desarrollan tecnología de chips de ADN
- Affymetrix
- Agilent Technologies
- CombiMatrix
- Eppendorf
- febit
- GE Healthcare (formalmente Amersham plc)
- GenePix
- Illumina
- Nanogen
- Nimblegen Systems
- Ocimum Biosolutions (adquirida por MWG Biotech)
- Roche Diagnostics
Enlaces externos
- DNA Microarrays in Health Care and Drug Discovery
- Microarray; How Is Working? (Animación)
- PLoS Biology Primer: Microarray Analysis
- Design DNA microarray probes
- GENIOM®-The Instrument: Design, production and application of custom microarrays in the researcher’s lab today
- Leming Shi's Genome Chip Resources
- Nature Genetics Free Issue on Gene Chips
- DNA Microarray Methodology
- How to build your own arrayer
- DNA microarray and DNA analysis technology
- How to build your own ink jet microarrayer
- Microarray protocols, how-to documents, free software
- Microarrayer tools and resources
- Rundown of microarray technology
- Large-Scale Gene Expression and Microarray Links and Resources
- Microarray data analysis
- Microarray Data Classification Server (MDCS)
- TiMAT: Tiling Microarray Analysis Tools
- The Microarray Gene Expression Data Society, and home of MIAME
- Try DNA microarray yourself in an interactive demonstration
- ArrayExpress at the European Bioinformatics Institute
- Gene Expression Omnibus (GEO) at NCBI
- Genevestigator at ETH Zurich
- Center for Functional Genomics, SUNY Albany's core lab providing microarray and related services to all
- The Science Creative Quarterly's overview of Microarrays - also excellent free hi-res schematic images available on the technique itself.
- Unidad de Genomica PCM-UCM. Servicio de Microarrays
Referencias
- ↑ ^ Schena M, Shalon D, Davis RW, Brown PO (1995). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science 270 (5235): 467–470. doi:10.1126/science.270.5235.467. PMID 7569999.
- ↑ ^ Lashkari DA, DeRisi JL, McCusker JH, Namath AF, Gentile C, Hwang SY, Brown PO, Davis RW (1997). "Yeast microarrays for genome wide parallel genetic and gene expression analysis". Proc Natl Acad Sci USA 94 (24): 13057–13062. doi:10.1073/pnas.94.24.13057. PMID 9371799.
Categorías:- Técnicas analíticas en biología molecular
- Expresión génica
- Bioinformática
Wikimedia foundation. 2010.