- Oligonucleótido
-
Un oligonucleótido es una secuencia corta de ADN o ARN, con cincuenta pares de bases o menos.
Tienen distintas funciones: se utilizan como cebadores en reacciones de amplificación, como sondas de hibridación y en bloqueos específicos de ARN mensajero.
Contenido
Síntesis de oligonucleótidos
Un oligonucleótido es una molécula compleja formada a su vez por varios nucleótidos, cada uno de los cuales está compuesto por una base nitrogenada, un hidrato de carbono y un grupo fosfato. La síntesis de oligonucleótidos se basa en una serie de reacciones en las que se van protegiendo y desprotegiendo cíclicamente los diferentes centros reactivos de la molécula:
- Protector del hidroxilo 2': Tert-butyl dimetilsilil (TBDMS).
- Protector del fosfato libre: B-cianoetilo.
- Protector de los heterociclos de las bases: diferentes grupos amida.
Aplicaciones en terapia génica
Los oligonucleótidos se emplean en terapia génica como estrategia para el silenciamiento de genes. Se utilizan oligonucleótidos de 12 a 20 pares de bases complementarios a los ARN mensajeros de los genes que queremos silenciar.
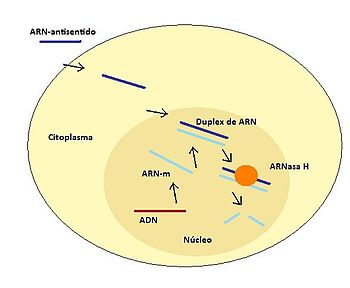
Dentro de la célula, el oligonucleótido se une a su ARN mensajero diana y bloquea su traducción, esto es, el ribosoma va a traducir el ARN mensajero hasta que se encuentra el oligonucleótido y el proceso se detiene. Además, el hecho de que la ARNasaH reconozca el híbrido oligo-ARN y lo degrade es otra ventaja de esta técnia. Es importante diseñar el oligonucleótido cerca del inicio de traducción o bien asegurarse de que el posible producto resultante no tenga función biológica, para que el silenciamiento sea efectivo.
Los oligonucleótidos ideales para terapia génica deben tener las siguientes características:
- Deben formar complejos estables con el material genético.
- Deben ser específicos de la diana.
- Deben poseer una alta estabilidad y una vida media más larga de lo habitual para resistir a las nucleasas.
- Deben poder pasar sin problemas a través de las membranas celulares.
- No deben acumularse en órganos u orgánulos celulares.
- Su tamaño debe ser de entre 18 y 20 pares de bases.
Los oligonucleótidos pueden usarse in vivo, pero poseen una vida media muy corta y una concentración baja. Precisamente porque no tiene efectos duraderos, el principal problema de los oligonucleótidos como terapia génica es que requieren dosis sucesivas.
Se modifican químicamente para tener una vida media más larga, bien en el oxígeno de los puentes fosfato, en las bases o en el fosfato que no se encuentra en los puntes de unión. Se consideran por tanto como una droga/medicamento, ya que tienen no alteran el ADN y terminan por degradarse.
Las posibles aplicaciones de los oligonucleótidos podrían darse en terapia antiviral, terapia antibacteriana, terapia antiparasitaria, terapia anticáncer ex vivo, tratamiento de enfermedades de la piel, inhibición de la inflamación, supresión de oncogenes o supresión de genes dominantes.
Oligonucleótidos para salto de exón
En las mutaciones que provocan un fin anticipado de la traducción, los oligonucleótidos se emplearían para inducir un procesamiento alternativo en el ARN mensajero mutante, sin inlcuir el exón que contiene la mutación. De esta manera, el codón stop prematuro no sería leído por el ribosoma y la proteína podría continuar traduciéndose, luego el producto final sería el mismo que el del gen normal, sin mutación.
Oligonucleótios catalíticos
Si se une al oligonucleótido una molécula de EDTA-Fe, esto provoca la destrucción activa del ARN mensajero al que se une, con lo que el oligonucleótido puede liberarse para degradar otro ARN mensajero.
Triple hélice
Un oligonucleótido también podría unirse a la doble hebra de ADN, dando lugar a una estructura conocida como triple hélice. Si se une a un gen concreto, estaría inhibiendo su transcripción, y por tanto, de nuevo, silenciándolo. Sólo se necesitarían dos oligonucleótidos por célula (uno por cada cromosoma del par), de modo que se evitaría el problema de la dosis. Esta técnica sería útil para mutaciones dominantes. Sin embargo, el superenrollamiento del ADN y su interacción con las histonas dificulta la entrada del oligonucleótido en la doble cadena.
Categorías:- Genética
- Nucleótidos
Wikimedia foundation. 2010.