- Haplogrupo R (ADNmt)
-
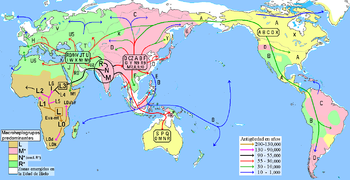
El haplogrupo R es un haplogrupo de ADN mitocondrial humano cuyos descendientes están esparcidos por todo Eurasia, Oceanía y América, siendo predominante en Eurasia Occidental, especialmente en Europa y con un promedio del 89%.[1]
Tiene una antigüedad aproximada de 60.000 a 65.000 años y probablemente se originó en el Subcontinente indio. Está definido por los marcadores genéticos 12705 y 16223.
Dada su diversidad, complejidad y extensión geográfica es considerado un macrohaplogrupo. Es derivado del haplogrupo N y cuenta entre sus descendientes a los haplogrupos B, F, H, V, J, T, P, U, K y numerosos subgrupos de R.
Contenido
Origen
La diversificación del haplogrupo R está relacionada -conjuntamente con los clados más antiguos de N y M- con la temprana expansión de la humanidad fuera de África. Según Macaulay et al. (2005)[2] esta expansión consistió en un solo proceso en el que primero aparecen N y M hace unos 63.000 años y luego el haplogrupo R divirgiendo rápidamente dentro de N hace unos 60.000 años.
Otros cálculos determinan que pudo originarse en el Sur de Asia apareciendo N hace unos 71.000 años y divirgiendo R en la misma región hace 67.000 (±14.000).[3]
En el Subcontinente Indio, R tiene amplia diversidad y gran antigüedad entre los diferenes grupos étnicos según sus status y entre las distintas familias lingüísticas. Entre las castas de la región Oeste de la India y entre las tribus de la región Sur, se observa mayor diversidad de haplogrupos que en otras regiones, sugiriendo así su origen y desarrollo autóctono.[4]
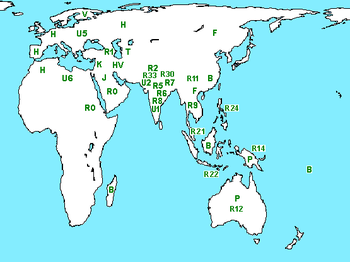
Clasificación de R* y su distribución
Los subgrupos del haplogrupo R y sus descendientes se resumen en el siguiente esquema:[5]
- Haplogrupo R (12705, 16223)
- R0 o pre-HV (73, 11719): Distribuído ampliamente. Frecuencia importante en kalashas (23%).[6] Se encuentra en la India, Pakistán, Irán, Arabia, Anatolia, Norte de África, Cuerno de África, y en Europa en Italia y Dalmacia. Se ha reportado presencia de R0 o HV en restos óseos de hace 24.000 años del hombre de cromañón en el norte de Italia.[7]
- R0a o pre-HV1: Común en Arabia y Medio Oriente. Menores frecuencias en Europa, Indostán y parte de África.
- HV (14766): Importantes frecuencias en kurdos, persas y gilekis.[6] Se encuentra en el Subcontinente Indio, Medio Oriente y Asia Central. A través de su clado más importante (H) es predominante en Europa y Eurasia Occidental en general.
- HV0 o pre-V
- V
- H
- HV0 o pre-V
- R1: Encontrado en India, Rusia, kurdos de Turkmenistán y Portugal.
- R2'JT o pre-JT (4216)
- R2: Importante en Baluchistán (Pakistán),[6] también se le encuentra en India, Irán y Georgia.
- JT: A través de sus descendientes J y T es típico de todo Eurasia Occidental, común en Europa, Medio Oriente, Siberia Occidental, Subcontinente Indio y África del Norte.
- R5: Presente en el Subcontinente Indio ampliamente disperso. Especialmente en Madhya Pradesh (India) con 17%.[8]
- R6'7 (16362) Especialmente en hablantes de lenguas austroasiáticas de la India.[9]
- R6: Típico del Subcontinente Indio.[10]
- R7: En el Subcontinente Indio, especialmente en Bihar (ver mapa).
- R8: En Megalaya, India.
- (16304)
- R9 (3970, 13928C): (Subclado R9a está reclasificado como F3)
- R9b: Común en el Sudeste de Asia.[11] Encontrado en todo Indonesia, en Vietnam, Malasia, en nativos malayos como los Semelai con 28%.[12]
- R9c'F (249d)
- R9c: Ampliamente disperso en Asia Oriental y Sudeste de Asia. Destacan los negritos Batak (Palawan, Filipinas) con 58%[13] y Alor (Indonesia) con 11%.[14]
- F: Típico del Asia Oriental y Sudeste de Asia, con las frecuencias más altas en los aborígenes de Taiwán y en Guangdong (China). Bajas frecuencias en el Subcontinente Indio.
- R22: Típico de Indonesia, especialmente en las islas menores de la Sonda (8%).[14] Poco en Tailandia.
- R9 (3970, 13928C): (Subclado R9a está reclasificado como F3)
- R11'B (16189)
- R11: Encontrado en China, en especial en los lahu de Yunnan con 12.5%;[15] también en Japón y Rajastán (India).
- B: Muy extendido en todo América originaria, Asia Oriental, Sudeste de Asia, Polinesia, Micronesia y Melanesia. Bajas frecuencias en Asia Meridional y Sur de Siberia.
- R24: Encontrado en Filipinas.[16]
- R12'21
- R14: En Papúa Nueva Guinea[18] e islas Nicobar.
- R23: En Bali, Indonesia.[14]
- R30: Disperso en varios estados de la India.
- R31: Pequeño haplogrupo encontrado en la India (Andhra Pradesh, Uttar Pradesh, Rajastán).
- P: Característico de los aborígenes de Oceanía. Típico de Australia y Melanesia.
- U: A veces denominado UK o Uk por la inclusión del clado K. Es característico de todo Eurasia Occidental, común en Europa, Medio Oriente, Siberia Occidental, Subcontinente Indio y África del Norte. Es predominante en los lapones y pequeñas frecuencias en Oriente.
- U1
- U5
- U6
- U2'3'4'7'8'9
- U8
- K
- U8
- R0 o pre-HV (73, 11719): Distribuído ampliamente. Frecuencia importante en kalashas (23%).[6] Se encuentra en la India, Pakistán, Irán, Arabia, Anatolia, Norte de África, Cuerno de África, y en Europa en Italia y Dalmacia. Se ha reportado presencia de R0 o HV en restos óseos de hace 24.000 años del hombre de cromañón en el norte de Italia.[7]
Véase también
Eva mitocondrial (L) L0 L1-6 L1 L2 L3 L4 L5 L6 M N CZ D E G Q A S R I W X Y C Z B F R0 JT P U HV J T K H V Enlaces externos
- Árbol filogenético de R de van Oven M & Kayser M. 2009
- Macrohaplogrupo R* de Ian Logan
- Dispersión del Haplogrupo R, de la National Geographic
Referencias
- ↑ Eupedia.com, Distribution of European mitochondrial DNA (mtDNA) haplogroups by region in percentage
- ↑ Vincent Macaulay et al 2005, Single, Rapid Coastal Settlement of Asia Revealed by Analysis of Complete Mitochondrial Genomes, Science 1034-1036 DOI: 10.1126/science.1109792
- ↑ Pedro Soares et al 2009, Correcting for Purifying Selection: An Improved Human Mitochondrial Molecular Clock. y su página suplemento The American Journal of Human Genetics, Volume 84, Issue 6, 740-759, 04 June 2009
- ↑ Suvendu Maji et al 2008, Distribution of Mitochondrial DNA Macrohaplogroup N in India with Special Reference to Haplogroup R and its Sub-Haplogroup U Int J Hum Genet, 8(1-2): 85-96 (2008)
- ↑ van Oven M, Kayser M. 2009. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. Hum Mutat 30(2):E386-E394. http://www.phylotree.org, mtDNA tree Build 2 (14 Oct 2008)
- ↑ a b c Lluís Quintana-Murci et al. 2004, Where West Meets East: The Complex mtDNA Landscape of the Southwest and Central Asian Corridor. Am. J. Hum. Genet. 74:000–000, 2004
- ↑ David Caramelli et al. 2003, Evidence for a genetic discontinuity between Neandertals and 24,000-year-old anatomically modern Europeans. University of Utah, Salt Lake City, UT, and approved March 27, 2003
- ↑ Mait Metspalu et al. 2005, Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans. BMC Genetics 2004, 5:26
- ↑ Gyaneshwer Chaubey et al. 2008 Phylogeography of mtDNA haplogroup R7 in the Indian peninsula BMC Evolutionary Biology 2008, 8:227
- ↑ Suvendu Maji et al. 2008, Distribution of Mitochondrial DNA Macrohaplogroup N in India with Special Reference to Haplogroup R and its Sub-Haplogroup U. Int J Hum Genet, 8(1-2): 85-96 (2008)
- ↑ Haplogrupo R9b, Web del ADNmt de Ian Logan
- ↑ a b Catherine Hill et al. 2006, Phylogeography and Ethnogenesis of Aboriginal Southeast Asians Mol. Biol. Evol. 23(12):2480–2491. 2006
- ↑ Clarissa Scholes et al. Genetic Diversity and Evidence for Population Admixture in Batak Negritos from Palawan, AMERICAN JOURNAL OF PHYSICAL ANTHROPOLOGY. 000:000–000 (2011)
- ↑ a b c C. Hill et al. 2007, A Mitochondrial Stratigraphy for Island Southeast Asia. Am J Hum Genet. 2007 January; 80(1): 29–43.
- ↑ Masashi Tanaka et al. 2004, Mitochondrial Genome Variation in Eastern Asia and the Peopling of Japan. Genome Res. 2004. 14: 1832-1850
- ↑ Tabbada, Kristina. et al. 2009, Philippine mitochondrial DNA diversity: a populated viaduct between Taiwan and Indonesia? Molecular Biology and Evolution, doi:10.1093/molbev/msp215
- ↑ Melanie J. Pierson et al. 2006, Deciphering Past Human Population Movements in Oceania: Provably Optimal Trees of 127 mtDNA Genomes. MBE Advance Access published July 19, 2006
- ↑ Haplogrupo R14, Web del ADNmt de Ian Logan
.
Categoría:- Haplogrupos mitocondriales humanos
- Haplogrupo R (12705, 16223)
Wikimedia foundation. 2010.