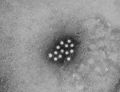
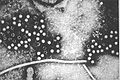
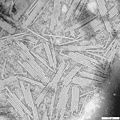
- Virus ARN monocatenario positivo
-
Virus ARN monocatenario positivo

CalicivirusClasificación de los virus Grupo: IV (Virus ARN monocatenario positivo) Órdenes, familias y géneros - Orden Nidovirales
- Familia Arteriviridae
- Familia Coronaviridae
- Familia Roniviridae
- Orden Picornavirales
- Familia Comoviridae
- Familia Dicistroviridae
- Familia Marnaviridae
- Familia Picornaviridae
- Familia Sequiviridae
- Familias sin asignar
- Familia Astroviridae
- Familia Barnaviridae
- Familia Bromoviridae
- Familia Caliciviridae
- Familia Closteroviridae
- Familia Flaviviridae
- Familia Flexiviridae
- Familia Leviviridae
- Familia Luteoviridae
- Familia Narnaviridae
- Familia Nodaviridae
- Familia Potyviridae
- Familia Tetraviridae
- Familia Togaviridae
- Familia Tombusviridae
- Familia Tymoviridae
- Géneros sin clasificar
- Género Benyvirus
- Género Cheravirus
- Género Furovirus
- Género Hepevirus
- Género Hordeivirus
- Género Idaeovirus
- Género Iflavirus
- Género Ourmiavirus
- Género Pecluvirus
- Género Pomovirus
- Género Sadwavirus
- Género Sobemovirus
- Género Tobamovirus
- Género Tobravirus
- Género Umbravirus
Un virus ARN monocatenario positivo (o virus (+)ssRNA) es un virus que tiene ácido ribonucleico (ARN) de cadena sencilla de sentido positivo como material genético y no se replica usando ADN intermedio. Pertenecen al Grupo IV de la clasificación de Baltimore.[1] [2] Es un grupo de virus del tipo ARN monocatenario, los cuales pueden clasificarse según el sentido o polaridad de su ARN en negativos o positivos. Los virus ARN positivos son idénticos al ARNm viral y por lo tanto pueden ser inmediatamente traducidos por la célula huésped. Aunque el ARN purificado de un virus positivo puede causar directamente una infección, seguramente sea menos infeccioso que el virus completo. La replicación tiene lugar principalmente en el citoplasma y no es tan dependiente del ciclo celular como en los virus ADN.
Contenido
Multiplicación
Los virus ARN de sentido positivo tienen genomas con la misma polaridad del ARNm y pueden ser empleados directamente para la síntesis de proteínas usando la maquinaria de traducción de la célula huésped. Una de estas proteínas codificadas es la ARN replicasa, una ARN polimerasa que copia el ARN viral sin necesidad de pasar por una cadena de ADN intermedia.
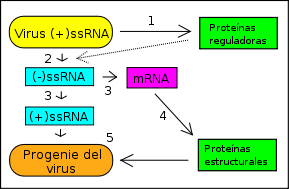
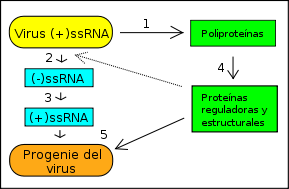
Síntesis de proteínas: (+)ssRNA (=mRNA) → proteínas Replicación del genoma: (+)ssRNA → (-)ssRNA → (+)ssRNA Enzimas: RNA polimerasas codificadas por el virus: (+)ssRNA → (-)ssRNA, (-)ssRNA → (+)ssRNA Por tanto, la expresión genética de un virus ARN monocatenario positivo comienza con la traducción más que con la transcripción. Durante la traducción han de formarse varias proteínas a partir de la cadena de ARN inicial y dependiendo como se formen las distintas proteínas, podemos distinguir dos tipos de virus:[3]
- Virus que generan cadenas de ARNm subgenómicas durante el ciclo replicativo.
- Virus en los cuales el genoma representa el único ARNm viral y la expresión es regulada principalmente a nivel traduccional y por la proteólisis limitada de poliproteínas.
En el primero de los casos, la multiplicación del virus comprende las siguientes etapas:
- Traducción temprana del ARN como si fuese ARNm y obtención de las proteínas tempranas (reguladoras), entre ellas la ARN replicasa.
- Síntesis del ARN monocatenario negativo a partir del molde de ARN monocatenario positivo por la ARN polimerasa y formación del complejo replicativo. El ARN monocatenario negativo no se libera, sino que permanece siempre asociado al complejo replicativo.
- El complejo replicativo realiza la síntesis de ARN monocatenario positivo, ARNm y ARN monocatenario negativo.
- Traducción tardía del ARN monocatenario positivo y ARNm y obtención de las proteínas tardías (estructurales), que probablemente fuerzan al complejo replicativo a producir un mayor porcentaje de ARN monocatenario positivo.
- Ensamblado de las proteínas estructurales y del ARN monocatenario positivo y maduración de los viriones.
En el caso en que no se generen cadenas de ARNm subgenómico, la expresión es regulada principalmente a nivel traduccional y por la proteólisis limitada de poliproteínas. De esta forma se producen varias proteínas a partir de la misma cadena de ARN.
El genoma puede ser no segmentado con un único ORF (Potyviridae, Picornaviridae y Sequiviridae), no segmentado con varios ORF (Togaviridae, Caliciviridae y Tobamovirus), dos segmentos con un único ORF (Comoviridae, Nodaviridae, Tetraviridae y Bymovirus), dos segmentos con varios ORF (Tobravirus, Furovirus y Enamovirus) o tres segmentos con varios ORF (Hordeivirus y Bromoviridae). El tamaño del genoma está comprendido entre menos de 5 y 30 kb.
Especies
Estos virus pueden infectar vertebrados (Astroviridae, Caliciviridae, Coronaviridae, Flaviviridae, Picornaviridae, Togaviridae y Arterivirus), insectos (Nodaviridae y Tetraviridae), plantas (Bromoviridae, Comoviridae, Potyviridae, Sequiviridae, Tombusviridae y varios géneros no asignados), hongos (Barnaviridae) o bacterias (Leviviridae). Entre los virus de este grupo que afectan a los seres humanos, destacan SARS, fiebre amarilla, virus del Nilo Occidental, hepatitis C, dengue, poliomielitis, resfriado común, rubéola, hepatitis A, hepatitis E. De importancia en la agricultura es el mosaico del tabaco.
Referencias
- ↑ "ICTVdb Index of Viruses: Virus Taxonomy, 8th Reports of the International Committee on Taxonomy of Viruses: Listing in Taxonomic Order." (Website). U.S. National Center for Biotechnology Information, National Library for Medicine, National Institutes of Health. Consultado el 09-28-2007.
- ↑ Virus Taxonomy 2008, International Committee on Taxonomy of Viruses (website). Consultado el 14-11-2008.
- ↑ M.A. Tijms, L.C. van Dinten, A.E. Gorbalenya y E.J. Snijder (2001) A zinc finger-containing papain-like protease couples subgenomic mRNA synthesis to genome translation in a positive-stranded RNA virus, PNAS. February 13, 2001, vol. 98, no. 4, pp. 1889--1894.
Véase también
Enlaces externos
- Orden Nidovirales
Wikimedia foundation. 2010.