- NADH deshidrogenasa
-
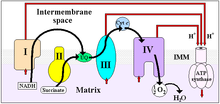
La NADH deshidrogenasa, NADH:ubiquinona oxidorreductasa (EC 1.6.5.3) o complejo I es un gran complejo multienzimático que cataliza la transferencia de electrones del NADH al coenzima Q en la cadena respiratoria.
Es el mayor complejo de la cadena respiratoria; en los mamíferos consta de 45 cadenas polipeptídicas, de las cuales, siete están codificadas por el genoma mitocondrial. Contiene FMN como grupo prostético y 8 cúmulos hierro-azufre.
Su estructura tienen forma de "L" con un gran dominio en la membrana (con alrededor de 60 hélices transmembrana) y un dominio periférico hidrófilo donde se produce la reducción del NADH.
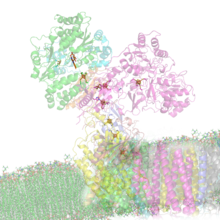 Estructura de la NADH deshidrogenasa; la membrana interna mitocondrial queda en la parte inferior de la figura.
Estructura de la NADH deshidrogenasa; la membrana interna mitocondrial queda en la parte inferior de la figura.
Constituye el punto de entrada a la cadena de transporte electrónico en las bacterias y en la membrana interna de las mitocondrias de las células eucariota. La transferencia electrónica se inicia mediante la oxidación del NADH ya que sus dos electrones son transferidos simultáneamente a un FMN unido no covalentemente a la enzima, y desde allí, a través de una serie de clusters Fe-S (transportadores de electrones capaces de aceptar un electrón por ciclo) al aceptor final, la coenzima Q (ubiquinona, transportador electrónico liposoluble) o a aceptores artificiales como ferrocianuro o rutenio (III) hexaamina.[1] [2]
Simultáneamente al transporte electrónico, el complejo I bombea protones a través de la membrana, contribuyendo así a la generación de un gradiente electroquímico. La estequiometría aceptada para el bombeo de protones es 2H+/e-.[3] [4] Este proceso produce el 40% del gradiente transmembrana generado en la oxidación del NADH por la cadena respiratoria mitocondrial.[5] Recientemente se demostró que en ciertos procariotas el complejo transloca cationes Na+ en lugar de protones y que de este modo se genera una fuerza sodio-motriz en lugar de protón-motriz.[6] [7]
Más de cien genes están involucrados en la biosíntesis del complejo I, participan en: transcripción, traducción, transporte, procesamiento, inserción de cofactores y ensamblado. El proceso de biosíntesis debe estar estrictamente regulado para mantener los niveles de actividad metabólica en células y tejidos, de acuerdo a su demanda energética.[3] [4]
Tradicionalmente se ha denominado como complejo I a la enzima mitocondrial mientras que su contraparte bacteriana se conoce como NADH deshidrogenasa tipo I.[8] [9]
Patología
Mutaciones en las subunidades del complejo I pueden causar enfermedades mitocondriales, entre las que destaca el síndrome de Leigh.
Hay algunas evidencias de que defectos en el complejo I pueden jugar cierto papel en la etiología de la enfermedad de Parkinson, tal vez porque el complejo I (al igual que el III) pueden padecer fugas de electrones hacia el oxígeno formando superóxidos muy tóxicos).
Referencias
- ↑ Carroll J, Fearnley IM, Shannon RJ, Hirst J, Walker JE: Analysis of the subunit composition of complex I from bovine heart mitochondria. Mol Cell Proteomics 2003, 2:117-126.
- ↑ Dupuis A, Prieur I, Lunardi J: Toward a characterization of the connecting module of complex I. J Bioenerg Biomembr 2001, 33:159-168.
- ↑ a b Schultz BE, Chan SI: Structures and proton-pumping strategies of mitochondrial respiratory enzymes. Ann Rev Biophys Biomol Struct 2001, 30:23-65.
- ↑ a b Yano T: The energy-transducing NADH: quinone oxidoreductase, complex I. Mol Aspects Med 2002, 23:345-368.
- ↑ Hinchliffe P, Sazanov LA: Organization of iron-sulfur clusters in respiratory complex I. Science 2005, 309:771-774.
- ↑ Friedrich T, Bottcher B: The gross structure of the respiratory complex I: a Lego System. Biochim Biophys Acta 2004, 1608:1-9.
- ↑ Hayashi M, Nakayama Y, Unemoto T: Recent progress in the Na(+)-translocating NADH-quinone reductase from the marine Vibrio alginolyticus. Biochim Biophys Acta 2001, 1505:37-44.
- ↑ Hatefi Y: The mitochondrial electron transport and oxidative phosphorylation system. Ann Rev Biochem 1985, 54:1015-1069.
- ↑ Matsushita K, Ohnishi T, Kaback HR: NADH-ubiquinone oxidoreductases of the Escherichia coli aerobic respiratory chain. Biochemistry 1987, 26:7732-7737.
Categoría:- Enzimas
Wikimedia foundation. 2010.