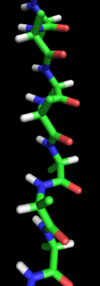
- Lámina alfa
-
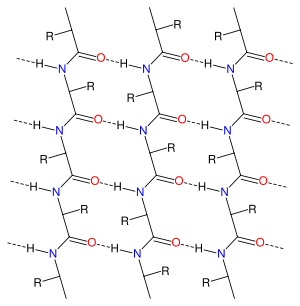 Diagrama de los patrones de los enlaces de hidrógeno en la estructura de lámina alfa. Los átomos de oxígeno se muestran en rojo y los de nitrógeno en azul; las líneas punteadas representan los enlaces de hidrógeno. Los grupos R representan la cadena lateral de los aminoácidos.
Diagrama de los patrones de los enlaces de hidrógeno en la estructura de lámina alfa. Los átomos de oxígeno se muestran en rojo y los de nitrógeno en azul; las líneas punteadas representan los enlaces de hidrógeno. Los grupos R representan la cadena lateral de los aminoácidos.
La lámina alfa es una estructura secundaria hipotética de proteínas, la cual fue propuesta por vez primera por Linus Pauling y Robert Corey en 1951.[1] [2] [3] El patrón de puentes de hidrógeno en una lámina alfa es similar al de una lámina beta, pero la orientación de los grupos carbonilo y amino en el enlace peptídico es diferente; en una lámina alfa aislada, todos los grupos carbonilo están orientados en la misma dirección de un lado del plegamiento, y todos los grupos amino se encuentran orientados en la misma posición pero del lado opuesto de la lámina. Por lo tanto, la lámina alfa acumula una inherente separación de carga electrostática, con un borde de la lámina exponiendo los grupos carbonilo de carga negativa y el borde opuesto exponiendo los grupos de amino cargados positivamente. A diferencia de la hélice alfa y la lámina beta, la configuración de lámina alfa no requiere que todos los residuos de aminoácidos que la componen se encuentren dentro de los límites de una única región de ángulos diedros; en cambio, la lámina alfa contiene residuos de diedros alternando en las tradicionales regiones dextrógiras (αR) y levógiras (αL) helicoidales del espacio de Ramachandran. Aunque la lámina alfa es muy raramente observada en estructuras de proteínas naturales,[4] es una forma estable accesible a las proteínas amiloidogénicas en simulaciones de dinámica molecular.[5] Las láminas alfa también se han observado con cristalografía de rayos X de estructuras de péptidos diseñados.[4]
Evidencia experimental
Cuando Pauling y Corey, propusieron por primera vez la estructura de lámina alfa, sugirieron que estaba de acuerdo con los resultados de difracción en fibras de beta-queratina.[2] Sin embargo, ya que la lámina alfa no parecen ser favorables energéticamente, señalaron que más los más común sería que hubieran láminas beta entre las proteínas normales,[3] y la posterior demostración de que la beta-queratina está formada por láminas beta restaron importancia a la propuesta de láminas alfa. Recientemente, la conformación lámina alfa se ha observado en casos aislados en estado nativo en proteínas resueltas por cristalografía de rayos X o RMN de proteínas, aunque una lámina alfa extendida no se ha identificado en proteínas naturales conocidas. Algunas proteínas que en estado nativo poseen regiones o patrones de enlaces de hidrógeno en lámina alfa incluyen sinaptotagmina, lisozima, y canales de potasio, donde las láminas alfa se encuentran en el poro conductor del ion.[4]
Las conformaciones en lámina alfa se han observado en estructuras cristalizadas de péptidos cortos no naturales, especialmente aquellos que contienen una mezcla de aminoácidos L y D. La primera estructura de cristal observada conteniendo una lámina alfa fue el tripéptido Boc–AlaL–a-IleD–IleL–OMe.[6] Otros péptidos que forman estructuras de lámina alfa incluyen dipéptidos[7] y tripéptidos basados en difenil-glicina.[8]
Papel en la amiloidogénesis
La lámina alfa ha sido propuesto como un posible estado intermedio en el cambio conformacional en la formación de fibras de amiloide por péptidos y proteínas como la beta amiloide, repeticiones de poliglutamina, lisozima, proteínas priónicas y repeticiones de transtiretina, todos los cuales están asociados con enfermedades por mal plegamiento de proteínas. Por ejemplo, el beta amiloide es un importante componente de las placas de amiloide en el cerebro de pacientes con la enfermedad de Alzheimer,[5] y repeticiones de poliglutamina en la proteína huntingtina asociada con la enfermedad de Huntington.[9] Estas proteínas se someten a un cambio conformacional desde estructuras como un ovillo aleatorio o una hélice alfa a estructuras muy ordenadas como una lámina beta encontrada en fibras amiloides. La mayoría de las láminas beta en proteínas conocidas se encuentran "retorcidas" alrededor de 15° para obtener óptimos enlaces de hidrógeno y empaquetamiento estérico, sin embargo, algunas pruebas de cristalografía de electrones indica que al menos algunas fibras amiloide pueden contener láminas "planas" con con sólo 1-2,5° de giro.[10] Un intermediario de amiloide en la conformación de lámina alfa se sugiere para explicar algunas características anómalas del proceso de formación de fibras de amiloide, como la evidente dependencia de la secuencia de aminoácidos de la amiloidogénesis a pesar de la creencia de que el plegamiento de amiloide se estabiliza por el propio esqueleto peptídico de la proteína y la aceleración en el proceso de formación de fibras en presencia de un campo magnético.[11] [12]
Xu,[13] utilizando microscopía de fuerza atómica, demostró que la formación de fibras amiloides es un proceso de dos etapas en las que las proteínas primero se agregan en esperas coloidales de unos ~ 20 nm de diámetro. A continuación, las esferas se unen espontáneamente para formar cadenas lineales, que evolucionan en fibras amiloides maduras. La formación de estas cadenas lineales parece estar motivada por el desarrollo de un dipolo electrostático en cada una de las esferas coloidales lo suficientemente fuerte como para superar la repulsión de Coulomb. Esto sugiere un posible mecanismo por el cual la lámina alfa puede promover la agregación amiloide; el enlace peptídico tiene un relativamente grande dipolo electrostático intrínseca, pero normalmente los dipolos de enlaces peptídico cercanos se anulan unos a otros. En la lámina alfa, a diferencia de otras conformaciones, los enlaces péptidicos está orientados en paralelo a fin de que los dipolos de los enlaces individuales puedan sumarse para crear un fuerte dipolo electrostático global.
La proteína lisozima es una de los pocas proteínas que en estado nativo contiene una región de lámina alfa; tanto en pollos como en humanos presenta una estructura en lámina alfa cerca del sitio de una mutación genética conocida que se sabe que causa amiloidosis hereditaria en los humanos, generalmente una enfermedad genética autosómica dominante.[4] Las simulaciones de dinámica molecular de la proteína mutante revelan que la región alrededor de la mutación posee una conformación de lámina alfa.[5] La lisozima es una de las proteínas naturales conocidas que forma fibras amiloides en condiciones experimentales, y tanto la región en lámina alfa nativa y el sitio de la mutación caen en una región mayor identificada como el núcleo de la formación de fibras amiloides.[14] [15]
Se ha sugerido un mecanismo directo para la interconversión entre lámina alfa y lámina beta, sobre la base de péptidos girando en el planto en donde el dipéptido αRαL se invierte para producir un ángulo diedro de conformación ββ. Este proceso también se ha observado en las simulaciones de transtiretina[16] e implicado en determinadas familias de proteínas observadas mediante el examen de su conformación en el ángulo diedro enestructuras cristalinas.[17]
Referencias
- ↑ Pauling, L. & Corey, R. B (1951). «The pleated sheet, a new layer configuration of polypeptide chains». Proc. Natl. Acad. Sci. USA 37: pp. 251–256. PMID 14834147.
- ↑ a b Pauling, L. & Corey, R. B. (1951). «The structure of feather rachis keratin». Proc. Natl. Acad. Sci. USA 37: pp. 256–261. PMID 14834148.
- ↑ a b Pauling, L. & Corey, R. B. (1951). «Configurations of Polypeptide Chains With Favored Orientations Around Single Bonds: Two New Pleated Sheets». Proc. Natl. Acad. Sci. USA 37: pp. 729–740. PMID 16578412.
- ↑ a b c d Daggett V. (2006). «Alpha-sheet: The toxic conformer in amyloid diseases?». Acc Chem Res 39 (9): pp. 594-602. PMID 16981675.
- ↑ a b c Armen RS, DeMarco ML, Alonso DO, Daggett V. (2004). «Pauling and Corey's alpha-pleated sheet structure may define the prefibrillar amyloidogenic intermediate in amyloid disease». Proc Natl Acad Sci USA 101 (32): pp. 11622-7. PMID 15280548.
- ↑ Di Blasio B, Saviano M, Fattorusso R, Lombardi A, Pedone C, Valle V, Lorenzi GP (1994). «A crystal structure with features of an antiparallel alpha-pleated sheet». Biopolymers 34 (11): pp. 1463-8. PMID 7827259.
- ↑ De Simone G, Lombardi A, Galdiero S, Nastri F, Di Costanzo L, Gohda S, Sano A, Yamada T, Pavone V (2000). «The crystal structure of a Dcp-containing peptide». Biopolymers 53 (2): pp. 182-8. PMID 10679622.
- ↑ Pavone V, Lombardi A, Saviano M, Nastri F, Zaccaro L, Maglio O, Pedone C, Omote Y, Yamanaka Y, Yamada T. (1998). «Conformational behaviour of C(alpha,alpha)-diphenylglycine: folded vs. extended structures in DphiG-containing tripeptides». J. Pept. Sci. 4 (1): pp. 21-32. PMID 9523753.
- ↑ Armen RS, Bernard BM, Day R, Alonso DO, Daggett V (2005). «Characterization of a possible amyloidogenic precursor in glutamine-repeat neurodegenerative diseases». Proc Natl Acad Sci USA 102 (38): pp. 13433-8. PMID 16157882.
- ↑ Jimenez, J. L., Nettleton, E. J., Bouchard, M., Robinson, C. V., Dobson, C. M. & Saibil, H. R (2002). «The protofilament structure of insulin amyloid fibrils». Proc. Natl. Acad. Sci. USA 99 (14): pp. 9196–9201. PMID 12093917.
- ↑ Fraser, P. E., Duffy, L. K., O'Mally, M. B., Nguyen, J., Inouye, H. & Kirschner, D. A (1991). «Morphology and antibody recognition of synthetic beta-amyloid peptides». J. Neurosci. Res. 28: pp. 474–485. PMID 1908024.
- ↑ Malinchik, S. B., Inouye, H., Szumowski, K. E. & Kirschner, D. A. (1998). «Structural analysis of Alzheimer's beta(1-40) amyloid: protofilament assembly of tubular fibrils». Biophys. J. 74: pp. 537–545. PMID 9449354.
- ↑ Xu S. (2007). «Aggregation drives "misfolding" in protein amyloid fiber formation». Amyloid 14 (2): pp. 119-31. PMID 17577685.
- ↑ Frare D, Polverino de Laureto P, Zurdo J, Dobson CM, Fontana A. (2004). «A highly amyloidogenic region of hen lysozyme». J Mol Biol 340 (5): pp. 1153-65. PMID 15236974.
- ↑ Frare D, Mossuto MF, Polverino de Laureto P, Dumoulin M, Dobson CM, Fontana A. (2006). «Identification of the core structure of lysozyme amyloid fibrils by proteolysis». J Mol Biol 361 (3): pp. 551-61. PMID 16859705.
- ↑ Milner-White JE, Watson JD, Qi G, Hayward S. (2006). «Peptide plane can flip in two opposite directions: implication in amyloid formation of transthyretin». J Phys Chem B 110 (12): pp. 5829-33. PMID 16553385.
- ↑ Milner-White JE, Watson JD, Qi G, Hayward S. (2006). «Amyloid formation may involve alpha- to beta sheet interconversion via peptide plane flipping». Structure 17 (9): pp. 1369-76. PMID 16962968.
Wikimedia foundation. 2010.