- Proteína hierro-azufre
-
Proteína hierro-azufre
Las proteínas hierro-azufre son proteínas que se caracterizan por la presencia de centros hierro-azufre que contienen grupos sulfuro unidos a centros de dos a cuatro átomos de hierro en estados de oxidación variables. Los centros hierro-azufre se encuentran en una variedad de metaloproteínas, como la ferredoxina, NADH deshidrogenasa, hidrogenasas, coenzima Q-citocromo c reductasa, succinato-coenzima Q reductasa y nitrogenasa.[1] Los centros de hierro-azufre son más conocidos por su papel en las reacciones de oxidación-reducción en el transporte de electrones mitocondrial. Tanto el complejo I como el complejo II de la fosforilación oxidativa tienen múltiples centros Fe-S. También poseen muchas otras funciones, entre ellas la catálisis como en el caso de la aconitasa, la generación de radicales como en las enzimas dependientes de SAM, y como donantes de azufre en la biosíntesis del ácido lipoico y la biotina. Además, algunas proteínas Fe-S regulan la expresión génica. Las proteínas Fe-S son vulnerables al ataque del óxido nítrico biogénico.
Contenido
Estructura
En casi todas las proteínas Fe-S, los centros de Fe son tetraédricos y los ligandos son azufres de grupos tiolato de los residuos de cisteina. Los grupos sulfuro poseen dos o tres enlaces de coordinación. Son comunes tres diferentes tipos de centros Fe-S con estas características.
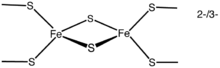
Centro 2Fe-2S
El más simple sistema polimetálico, el centro [Fe2S2], está constituido por dos iones de hierro enlazados a dos iones de azufre y coordinados por cuatro ligandos de cisteína (Fe2S2 en ferredoxinas) o por dos cisteínas y dos histidinas (en las proteínas de Rieske). Las proteínas oxidadas contienen dos iones Fe3+, mientras que la proteína reducida contiene solo un Fe3+ y un Fe2+. Estas especies existen en dos estados de oxidación, (FeIII)2 y FeIIIFeII.
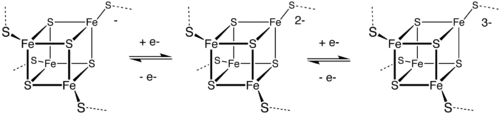
Centro 4Fe-4S
Una estructura común que consta de cuatro iones de hierro y cuatro de azufre ubicado en los vértices de una estructura en forma de cubo. Los centros de Fe son típicamente coordinados por ligandos de cisteína. Las proteínas transportadoras de electrones [Fe4S4] (como la ferredoxina) pueden ser a su vez subdivididas en ferredeoxinas de bajo-potencial (tipo bacteriano) y de alto potencial (HiPIP). Las ferredoxinas de bajo y alto potencial están relacionadas por el siguiente esquema redox:
El centro HiPIP, oscila entre [2Fe3+, 2Fe2+] (Fe4S42+) y [3Fe3+, Fe2+] (Fe4S43+). Los potenciales de este par redox varían de 0,4 a 0,1 V. En las ferrodoxinas bacterianas, el par de estados de oxidación es [Fe3+, 3Fe2+] (Fe4S4+) y [2Fe3+, 2Fe2+] (Fe4S42+). Los potenciales para este par redox varían de -0.3 a -0.7 V. Las dos familias de centros 4Fe-4S comparten el Fe4S42+. La diferencia en los pares redox es atribuida al grado de puentes de hidrógeno, que modifica fuertemente la basicidad de ligandos de cisteiníl tiolato. Otro par redox aún más reductor que la ferrodoxina bacteriana se encuentra en la nitrogenasa.
Algunos centros 4Fe-4S son capaces de unirse a sustratos y por ello son clasificados como enzimas. Por ejemplo, el centro Fe-S de la aconitasa, se une a aconitato con el centro de un ion de hierro carente del ligando tiolato. Este centro no sufre de una reacción redox, pero sirve como catalizador para convertir aconitato a isocitrato. En las enzimas dependientes de SAM, el centro se une y reduce la S-adenosilmetionina para generar un radical, el cuales está involucrado en varios procesos de biosíntesis.[2]
Centro 3Fe-4S
Existen varias proteínas que contienen centros [Fe3S4], las cuales presentan un ion de hierro menos que la forma de centros más común de tipo [Fe4S4]. Tres iones de azufre enlazan cada uno a dos de hierro, mientras que el cuarto azufre enlaza a tres iones de hierro. Sus estados de oxidación pueden variar desde [Fe3S4]+ (forma todo Fe3+) a [Fe3S4]2- (forma todo Fe2+). En algunas proteínas hierro azufre, el centro [Fe4S4] puede ser convertido reversiblemente por oxidación y pérdida de un ion de hierro a un centro [Fe3S4]. Por ejemplo la forma inactiva de la aconitasa posee un centro [Fe3S4] y es activada por la adición de Fe2+ y un agente reductor.
Otros centros Fe-S
Son comunes sistemas polimetálicos más complejos; por ejemplo los centros 8Fe y 7Fe en la nitrogenasa. La monóxido de carbono deshidrogenasa y la hidrogenasa-[FeFe] también presentan centros Fe-S inusuales.
Biosíntesis
La biosíntesis de los centros Fe-S es bien conocida.[3] [4] [5] En particular la biogénesis de los centros hierro-azufre ha sido estudiada en profundidad en la E. coli y A. vinelandii y en la levadura S. cerevisiae. Al menos tres sistemas biosínteticos han sido identificados, denominados sistemas nif, suf, e isc. El sistema nif es responsable de los centros de la enzima nitrogenasa. Los sistemas suf e isc son más genéricos siendo las proteínas relacionadas a isc las únicas presentes en el reino animal. El sistema isc ha sido estudiado mayormente en levaduras, donde varias proteínas constituyen la maquinaria biosintética a través de la ruta de isc. El proceso se lleva a cabo con el ensamblado del centro Fe/S en una proteína de andamiaje seguido por la transferencia de un centro preformado a la proteína destino. Este primer paso sucede en el citoplasma de los procariotas o en la mitocondria de los eucariotas. En organismos superiores los centros son transportados fuera de la mitocondria e incorporado en las enzimas extra-mitocondriales. Estos organismos también poseen un conjunto de proteínas involucradas en el transporte de centros Fe/S y procesos de incorporación que no son homólogos a los procesos encontrados en los sistemas de organismos procariotas.
Análogos sintéticos
Los análogos sintéticos de los centros de hierro-azufre encontrados en la naturaleza fueron descritos por vez primera por Richard H. Holm y colaboradores.[6] El tratamiento de sales de hierro con una mezcla de tiolatos y sulfuro permite crear derivados como el (↑ S. J. Lippard, J. M. Berg “Principles of Bioinorganic Chemistry” University Science Books: Mill Valley, CA; 1994. ISBN 0-935702-73-3.
- ↑ Susan C. Wang and Perry A. Frey (2007). «S-adenosylmethionine as an oxidant: the radical SAM superfamily» Trends in Biochemical Sciences. Vol. 32. p. 101. DOI 10.1016/j.tibs.2007.01.002.
- ↑ Johnson D, Dean DR, Smith AD, Johnson MK (2005). «247–281» Annual Review of Biochemistry. Vol. 74. DOI 10.1146/annurev.biochem.74.082803.133518.
- ↑ Johnson, M.K. and Smith, A.D. (2005) Iron–sulfur proteins in: Encyclopedia of Inorganic Chemistry (King, R.B., Ed.), 2nd edn, John Wiley & Sons, Chichester.
- ↑ Lill R, Mühlenho U (2005). «Iron–sulfur-protein biogenesis in eukaryotes» Trends in Biochemical Sciences. Vol. 30. pp. 133–141. DOI 10.1016/j.tibs.2005.01.006.
- ↑ T. Herskovitz, B. A. Averill, R. H. Holm, J. A. Ibers, W. D. Phillips and J. F. Weiher (1972). «Structure and Properties of a Synthetic Analogue of Bacterial Iron-Sulfur Proteins» Proceedings of the National Academy of Sciences. Vol. 69. n.º 9. pp. 2437–2441. DOI 10.1073/pnas.69.9.2437. PMID 4506765.
Categoría: Proteínas
Wikimedia foundation. 2010.