- Acetil-CoA carboxilasa
-
Acetil-CoA carboxilasa alfa[1] HUGO 84 Símbolo ACACA Símbolos alt. ACC1 Datos genéticos Código de gen ACACA Tipo de gen Gen codificante Locus Cr. 17 q21 Estructura/Función proteica Tamaño 2346 (aminoácidos) Bases de datos Número EC 6.4.1.2 Entrez 31 OMIM 200350 UniProt Q13085 Acetil-CoA carboxilasa beta[2] HUGO 85 Símbolo ACACB Símbolos alt. ACC-beta Datos genéticos Código de gen ACACB Tipo de gen Gen codificante Locus Cr. 12 q24.11 Estructura/Función proteica Tamaño 2458 (aminoácidos) Bases de datos Número EC 6.4.1.2 Entrez 32 OMIM 601557 UniProt O00763
La acetil-CoA carboxilasa ACAC (EC 6.4.1.2) es una enzima que cataliza la reacción de adición de un grupo bicarbonato al acetato para obtener malonato. Esta reacción consume una molécula de ATP.[3]- Acetil-CoA + HCO3- + ATP
 Malonil-CoA + ADP + fosfato
Malonil-CoA + ADP + fosfato
Esta enzima regula la biosíntesis de los ácidos grasos y su oxidación.[4] Utiliza como cofactor biotina que se une a la enzima mediante un residuo de lisina.[5] Adicionalmente utiliza dos átomos de manganeso por subunidad. La enzima se presenta como monómero, homodímero y homotetrámero. Puede formar polímeros filamentosos.[6]
Contenido
Mecanismo y estructura
Las enzimas carboxilasas dependientes de la biotina llevan a cabo una reacción en dos etapas. La enzima unida a la biotina es primero carboxilada por bicarbonato y ATP, y el grupo carboxilo unido temporalmente a la biotina es transferido a un sustrato aceptor como el piruvato o el acetil-CoA.[7]
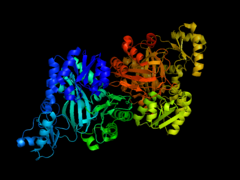
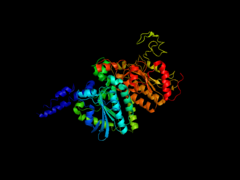
Estructuras cristalográficas de la acetil-CoA carboxilasa de la Escherichia coli. Figura 1. Estructura del dominio biotina carboxilasa de la ACAC de la E.Coli.Figura 2. Estructura del dominio carboxiltransferasa de la ACAC de la E.Coli.La primera etapa es mediada por el dominio biotina carboxilasa (BC) (EC 6.3.4.14) común a todas las carboxilasas dependientes de la biotina. El dominio BC puede ser dividido en tres subdominios (N-terminal, central y C-terminal). La región N-terminal proporciona parte del sitio activo; la región central corresponde al dominio de unión del ATP que es común en muchas enzimas dependientes del ATP que participan en la síntesis de macromoléculas. Por último, el subdominio C-terminal participa en la formación del multímero de enzimas.[7]
La segunda etapa de la reacción es realizada por el dominio carboxiltransferasa. Las regiones N- y C-terminal de este dominio comparten estructuras similares con una superhélice β-β-α central. La molécula de coenzima A se asocia con el subdominio N-terminal. En las acetil-CoA carboxilasas bacterianas los subdominios N- y C-terminal son codificados por dos polipéptidos diferentes.[4]
Isozimas
En el ser humano existen dos isozimas de la acetil-CoA carboxilasa llamadas alfa (ACACA) y beta (ACACB). La isozima alfa participa en la biogénesis de los ácidos grasos de cadena larga mientras que la isozima beta participa en la provisión de malonil-CoA y en la regulación de la oxidación de los ácidos grasos.[6] [8]
La isozima alfa se expresa en el cerebro, placenta, músculo esquelético, riñones, páncreas y tejidos adiposos. Se expresa en un bajo nivel en los tejidos pulmonares. No se ha detectado en el hígado.[6] La isozima beta se expresa predominantemente en el corazón, músculo esquelético e hígado.[8]
Los defectos en ACACA son causa de la deficiencia en acetil-CoA carboxilasa 1, también conocida como deficiencia en ACAC. Es una deficiencia innata en la síntesis de ácidos grasos asociada con daño cerebral severo, miopatía persistente y crecimiento pobre.[6]
Regulación
La regulación de la acetil-CoA carboxilasa es compleja, ya que se tienen que controlar los procesos de inhibición de la beta oxidación y la activación de la biosíntesis de lípidos.
Las ACACA y ACACB son reguladas transcripcionalmente por muchos promotores que median en la abundancia de la ACAC en respuesta al estado nutricional de las células. La activación de la expresión del gen a través de diferentes promotores resulta en splicing alternativo; el significado fisiológico de la isoformas formadas permanece desconocido.[9] La sensibilidad al estado nutricional resulta del control de estos promotores por los factores de transcripción como el SREBP1c, controlado por la insulina en el nivel transcripcional, y el ChREBP, que incrementa la expresión en dietas de alto contenido en carbohidratos.[10] [11]
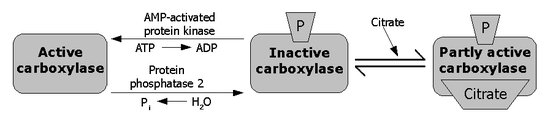
A través de un ciclo de retroalimentación, el citrato activa alostéricamente a la ACAC.[12] El citrato puede incrementar la polimerización de la ACAC para incrementar la actividad enzimática; aunque, no está claro si la polimerización es un mecanismo del citrato para incrementar la actividad de la ACAC o la polimerización es un resultado de los experimentos in vitro. Otros activadores alostéricos incluyen al glutamato y otros ácidos dicarboxílicos.[13] Las cadenas largas y cortas de acilos-CoA grasos son inhibidores de la ACAC.[14]
La fosforilación inhibitoria de la enzima puede ser resultado de la unión de las hormonas glucagón y epinefrina a los receptores de la superficie de la célula, pero la mayor causa de fosforilación es debida a un incremento en los niveles de AMP cuando el estatus de energía de la célula es bajo, resultando en la activación de la proteína kinasa activada por AMP (AMPK). AMPK es el principal regulador kinasa de la ACAC, capaz de fosforilar una serie de residuos serina en las dos isozimas.[15] En la ACACA, la AMPK fosforila Ser-79, Ser-1200 y Ser-1215. En la ACACB, la AMPK fosforila Ser-218.[16] La proteína kinasa A también tiene la habilidad de fosforilar la ACAC, con mucha mayor habilidad relativa para fosforilar la ACACB que la ACACA. De todas formas, el significado fisiológico de la proteína kinasa A en la regulación de la ACAC es todavía desconocido. Los investigadores creen que hay otras ACAC kinasas importantes para su regulación ya que hay muchos otros sitios de fosforilación posibles en la ACAC.[17]
Cuando la insulina se une a sus receptores situados en la membrana celular, activa una fosfatasa que defosforila la enzima causando la eliminación del efecto inhibitorio.
Referencias
- ↑ «ACACA Gene». Consultado el 22 de octubre de 2011.
- ↑ «ACACB Gene». Consultado el 22 de octubre de 2011.
- ↑ «ENZYME entry: EC 6.4.1.2». Consultado el 22 de octubre de 2011.
- ↑ a b «PROSITE documentation PDOC50980». Consultado el 22 de octubre de 2011.
- ↑ «PROSITE documentation PDOC00167». Consultado el 22 de octubre de 2011.
- ↑ a b c d «Acetyl-CoA carboxylase 1». Consultado el 22 de octubre de 2011.
- ↑ a b «PROSITE documentation PDOC50979». Consultado el 22 de octubre de 2011.
- ↑ a b «Acetyl-CoA carboxylase 2». Consultado el 22 de octubre de 2011.
- ↑ Barber MC, Price NT, Travers MT (March 2005). «Structure and regulation of acetyl-CoA carboxylase genes of metazoa». Biochim. Biophys. Acta 1733 (1): pp. 1–28. doi:. PMID 15749055.
- ↑ Field F. J., Born E., Murthy S. and Mathur S. N. (December 2002). «Polyunsaturated fatty acids decrease the expression of sterol regulatory element binding protein-1 in CaCo-2 cells: effect on fatty acid synthesis and triacylglycerol transport.». Biochem. J. 386 (Pt 3): pp. 855–64. doi:. PMID 12213084.
- ↑ Ishii S, Iizuka K, Miller BC, Uyeda K (October 2004). «Carbohydrate response element binding protein directly promotes lipogenic enzyme gene transcription». Proc Natl Acad Sci USA 101 (44): pp. 15597–602. doi:. PMID 15496471.
- ↑ Martin DB, Vagelos PR (June 1962). «The Mechanism of Tricarboxylic Acid Cycle Regulation of Fatty Acid Synthesis». J Biol Chem 237: pp. 1787–92. PMID 14470343.
- ↑ Boone AN, Chan A, Kulpa JE, Brownsey RW (April 2000). «Bimodal Activation of Acetyl-CoA Carboxylase by Glutamate». J Biol Chem 275 (15): pp. 10819–25. doi:. PMID 10753875.
- ↑ Faergeman NJ, Knudsen J (April 1997). «Role of long chain fatty acyl-CoA esters in the regulation of metabolism and in cell signalling». Biochem J. 323 (Pt 1): pp. 1–12. PMID 9173866.
- ↑ Park SH, Gammon SR, Knippers JD, Paulsen SR, Rubink DS, Winder WW (June 2002). «Phosphorylation-activity relationships of AMPK and acetyl-CoA carboxylase in muscle». J. Appl. Physiol. 92 (6): pp. 2475–82. doi:. PMID 12015362.
- ↑ Hardie DG (February 1992). «Regulation of fatty acid and cholesterol metabolism by the AMP-activated protein kinase». Biochim. Biophys. Acta 1123 (3): pp. 231–8. PMID 1536860.
- ↑ Brownsey RW, Boone AN, Elliott JE, Kulpa JE, Lee WM (April 2006). «Regulation of acetyl-CoA carboxylase». Biochem. Soc. Trans. 34 (Pt 2): pp. 223–7. doi:. PMID 16545081.
- Acetil-CoA + HCO3- + ATP
Wikimedia foundation. 2010.