- Multímero
-
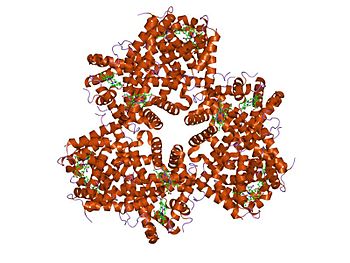 Multímero. Esta proteína es un multímero formado por tres cadenas polipeptídicas (subunidades proteicas), por lo tanto se trata de un trímero.
Multímero. Esta proteína es un multímero formado por tres cadenas polipeptídicas (subunidades proteicas), por lo tanto se trata de un trímero.
Un multímero es una proteína formada por varias subunidades proteicas, denominada oligómero cuando estas subunidades son pocas.[1]
De esta forma, un multímero es el nombre dado a una proteína que presenta estructura cuaternaria.
Términos relacionados con los multímeros
Muchas proteínas, especialmente aquellas con pesos moleculares elevados, presentan estructura cuaternaria, esto significa que están formadas por varias cadenas polipeptídicas (desde dos a centenares de ellas). Cada una de estas cadenas se denomina subunidad, y la unión de varias subunidades es lo que hemos llamado multímero, o proteína multisubunidad.[2] [1] [3]
En un complejo proteico estas subunidades pueden ser idénticas o bastante diferentes. Las proteínas con pocas subunidades se denominan oligómeros. Los oligómeros están formados por protómeros (unidades de repetición estructural en proteínas multiméricas que pueden estar formadas por una o varias subunidades). Un gran número de proteínas oligoméricas contienen dos o cuatro subunidades protoméricas, denominadas dímeros y tetrámetros respectivamente.[2] [1] [3]
Funciones de los multímeros[4]
La asociación de subunidades proteicas puede servir para una gran diversidad de funciones.
Reguladora
Muchos multímeros tienen funciones reguladoras; la unión de pequeñas moléculas puede alterar la interacción entre subunidades produciendo grandes cambios en la actividad de la proteína en respuesta a pequeños cambios en la concentración de sustrato o moléculas reguladoras. Las proteínas reguladoras impiden que se lleven a cabo reacciones metabólicas, por ejemplo, cuado éstas no sean necesarias (por ejemplo, no se sintetizarán ácidos grasos a partir de glucosa cuando la célula requiera mucho aporte energético. Todo esto está regulado por los moduladores o proteínas reguladoras, muchas son multímeros).
Mixta
En otros casos, las diferentes subunidades de un mimo multímero pueden llevar a cabo funciones separadas aunque relacionadas, tales como la catálisis y la regulación. Ésto resulta de gran utilidad para la célula, ya que se suprime la necesidad de tener dos proteínas (una para la regulación y otra para la catálisis) juntando todo el proceso en una única proteína multimérica. Así esta proteína podrá autoregular lareacción que catalice, ahorrando espacio y aumetando la eficiencia celular.
Estructural
Algunas asociaciones, como por ejemplo las proteínas fibrosas (colágeno) y las proteínas de cubierta de los virus, llamadas cápsides, tienen principalmente una función estructural, por tanto mantienen la conformación de la célula, y en l caso de los virus protegen la información genética del exteror.
Cadena de reacciones
Algunas estructuras proteicas son el sitio donde tienen lugar reacciones complejas con diversos pasos. Por ejemplo, cada uno de los ribosomas, lugar donde se produce la síntesis de proteínas, incorpora docenas de subunidades de proteínas junto con varias moléculas de RNA. Cada una de estas subunidades proteicas tiene una función específica en el proceso de la síntesis de proteínas: unión al mRNA, fijación de los tRNA, catalizador de la reacción de condensación para la formación del enlace peptídico entre aminoácidos, etc.[4]
Razones para la existencia de proteínas multiméricas (multímeros)
- La síntesis de subunidades aisladas es más eficaz que aumentar sustancialmente la longitud de una única cadena polipeptídica.
- En los complejos supramoleculares, como las fibras de colágeno, la sustitución de componentes más pequeños gastados o dañados puede realizarse de manera más eficaz.
- Las interacciones complejas de varias subunidades sirven para regular la función biológica de una proteína (Se obtiene, de esta manera,una autoregulación de las proteínas que catalizan reacciones metabólicas, por ejemplo).[2]
El multímero más conocido: La hemoglobina
La hemoglobina fue la primera proteína oligomérica de la que se determinó la estructura tridimensional. Está formada por cuatro cadenas polipeptídicas y cuatro grupos prostéticos hemo, en los que los átomos de hierro se encuentran en el estado ferroso (Fe2+). La parte proteica, la globina, está constituida por dos cadenas α (de 141 residuos cada una) y dos cadenas β (de 146 residuos cada una). Las letras griegas α y β no se refieren a estructuras secundarias.
En la derecha de la pantalla se puede apreciar la proteína hemoglobina con sus cuatro subunidades, y un átomo de hierro (Fe) en el interior de cada subunidad. Se trata de una mlécula de vital importancia ya que es la responsable del abastecimiento de oxígeno a las células del cuerpo.
La hemoglobina se concentra n los hematíes o glóbulos rojos, que son células sanguíneas compuestas por millones de hemoglobinas, y que, por tanto, permiten fijar millones y millones de moléculas de oxígeno provinente de los pulmones para abastecer las necesidades energéticas e las células del cuerpo.[3] [2]
Organización de las subunidades proteicas
Es importante destacar que las subunidades de los multímeros presentan generalmente (aunque no siempre) distintas organizaciones en formas específicas (patrones de simetría), dando lugar a simetría proteica.
Por ejemplo, las subunidades de hemoglobina están dispuestas en pares simétricos, con una subunidad α y una subunidad β cada uno.[5]
Tipos de multímeros
Existen dos tipos de multímeros: Aquellos que presentan asociación entre cadenas polipeptídicas idénticas o casi idénticas (homotípicos) y aquellos que presentan interacciones entre subunidades con estructuras muy distintas (heterotípicos).[4]
Multímeros homotípicos
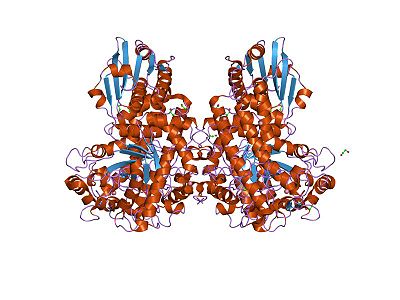 Multímero homotípico formado por dos subunidades proteicas idénticas (por tanto, un homodímero) que presenta simetría proteica cíclica.
Multímero homotípico formado por dos subunidades proteicas idénticas (por tanto, un homodímero) que presenta simetría proteica cíclica.
Éstos son aquellos cuyas subunidades son idénticas o casi idénticas.
Las subunidades de estos multímeros se mantienen unidas mediante los mismos enlaces que estabilizan la estructura terciaria y que se describirán más adelante: puentes salinos, enlaces de hidrógeno, fuerzas de Van der Waals, interacciones hidrófobas y a veces enlaces covalentes disulfuro.
Cada subunidad es una unidad asimétrica en el agregado, pero la estructura cuaternaria global de la proteína puede ofrecer una amplia variedad de simetría proteica en función de la geometría de las interacciones.
Un ejemplo de multímero homotípico sería la hemoglobina, que se ha descrito anteriormente.[4]
Cabe destacar que los multímeros homotípicos con únicamente dos subunidades se denominan homodímeros. En la imagen de la derecha podemos apreciar un multímero formado por dos subunidades iguales, por tanto un homodímero).[1]Multímeros heterotípicos
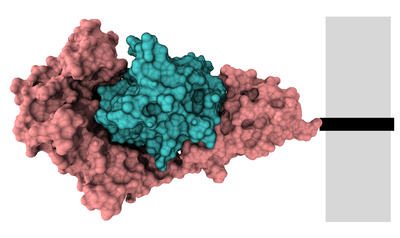 Proteína interleukina. Se trata de un multímero heterotípico formado por dos subunidades proteicas distintas (por tanto un heterodímero). No presenta simetría proteica.
Proteína interleukina. Se trata de un multímero heterotípico formado por dos subunidades proteicas distintas (por tanto un heterodímero). No presenta simetría proteica.
Éstos son los que tienen varias subunidades con formas diferentes. En ocasiones, pueden tener una docena o más subunidades distintas.
Las interacciones que mantienen unidas estas subunidades son del mismo tipo que las descritas para los multímeros homotípicos: fuerzas no covalentes (excepto enlaces covalentes disulfuro) en las superficies proteicas complementarias.
Un ejemplo sencillo de multímero heterotípico es el de la proteína BPTI, el inhibidor de la tripsina pancreática bovina. Recibe este nombre porque se une al centro activo de la enzima tripsina formando un complejo fuerte y específico, inhibiendo de este modo la actividad proteolítica de la tripsina en el páncreas. Esta unión es un ejemplo de multímero heterotípico ya que el complejo BPTI-tripsina esta formado por dos subunidades unidas fuertemente pero muy distintas estructuralmente.[4]
Hay que tener en cuenta que los multímeros heterotípicos que tienen sólo dos subunidades se denominan heterodímeros. En la imagen de la derecha podemos apreciar un multímero formado por dos subunidades de diferente estructura tridimensional (subunidades distintas), por tanto un heterodímero).[4]Ensamblaje (unión) de las subunidades de los multímeros[2]
Las subunidades polipeptídicas se ensamblan y se mantienen unidas por interacciones no covalentes, como:
- el efecto hidrófobo
Como con el plegamiento proteico, el efecto hidrófobo es claramente el más importante, ya que las superficies de unión complementarias entre las subunidades son semejantes a las observadas en el interior de los dominios de las proteínas globulares.
- los enlaces de hidrógeno
- encruzamientos covalentes
Aunque son menos numerosos, los encruzamientos covalentes estabilizan significativamente determinadas proteínas con varias subunidades.
Entre los ejemplos más destacados se encuentran los puentes disulfuro de las inmunoglobulinas y los enlaces de desmosina y lisinorleucina en determinadas proteínas del tejido conjuntivo. Los encruzamientos de desmosina conectan cuatro cadenas polipeptídicas en la proteína elastina del tejido conjuntivo, semejante a la goma. Se forman como consecuencia de diversas reacciones que implican la oxidación de las cadenas laterales de lisina.
Se produce un proceso semejante en la formación de lisinonorleucina, una estructura encruzada que se encuentra en la elastina y el colágeno.
Con mucha frecuencia, las interacciones entre las subunidades están afectadas por la unión de ligandos. En el alosterismo, el control de la función proteica mediante la unión de ligandos, la unión de un ligando a un lugar específico en una proteína, desencadena un cambio conformacional que altera su afinidad por otros ligandos. Los cambios conformacionales inducidos por el ligando en esas proteínas se denominan transiciones alostéricas, y los ligandos que las desencadenan efectores o moduladores.
Los efectos alostéricos pueden ser positivos o negativos, dependiendo de si la unión del modulador aumenta o disminuye la afinidad de la proteína por otros ligandos. Uno de los ejemplos de efecto alostérico mejor conocido es la unión reversible del O2 y otros ligandos a la hemoglobina. (Las enzimas alostéricas desempeñan un papel clave en el control de los procesos metabólicos)
Dinámica de las proteínas[2]
A pesar del énfasis dado hasta ahora a las fuerzas que estabilizan la estructura proteica, debe reconocerse que la función proteica requiere cierto grado de flexibilidad. El significado de la flexibilidad conformacional (fluctuaciones continuas, rápidas de la orientación precisa de los átomos en las proteínas) se ha descubierto al investigarse las interacciones proteína-ligando.
La función proteica suele implicar la apertura y el cerrado rápido de cavidades de la superficie de la molécula. La velocidad a la que las enzimas catalizan las reacciones está limitada en parte por la rapidez con la que pueden liberarse del lugar activo las moléculas producto. Cabe recordar que la transferencia de información entre las biomoléculas se produce cuando las moléculas con superficies complementarias precisas interaccionan en un proceso de interacciones no covalentes. La transferencia de información entre las moléculas siempre supone modificaciones de la estructura tridimensional.
Por ejemplo, las conformaciones de las subunidades de las moléculas de hemoglobina que unen O2 experimentan cambios estructurales específicos al unirse o separarse de las moléculas de oxígeno.Este ejemplo se observa en la animación de la derecha, donde, cuando aparece el término deoxy se visualiza la hemoglobina sin moléculas de O2, en cabio, cuando aparece el término oxy, se visualiza la hemoglobina oxigenada, el complejo hemoglobina-oxígeno. Sólo se visualiza la unión de un O2 ya que el lugar de unión del O2 con cada subunidad se encuentra e el interior de éstas (donde está la molécula de Fe).
Viendo la animación se aprecia la vital importancia que tiene el cambio de conformación de las proteínas que tiene lugar en la interacción con sus ligando s. Este cambio viene provocado por la simple unión del ligando a la proteína, ya que le induce un cambio de forma debido a la nueva formación de enlaces. Aunue éstos desaparecen una vez el ligando se desprende de la proteína, que vuelve a su forma inicial.
Este proceso de cambio conforacional se produce en muy poco tiempo (ya que se trata de nivel molecular), dando la posibilidad de que los enzimas catalicen millones de reacciones en muy poco tiempo. Así explicamos, por ejemplo, el rápido movimiento muscular. Éste se produce a causa de un gran número de microreacciones que provocan una contracción del sarcómero. De esta forma, el conjunto de contracciones de los sarcómeros dan lugar al movimiento tal y como lo conocemos. Se trata pues de reacciones moleculares llevadas a cabo por proteínas, a gran velocidad.
Por tanto, a nivel molecular es muy importante tener en cuenta la cinética enzimática para poder entender el funcionamiento de los seres vivos y su estructura.[2] [3]
Métodos para determinar si una proteína está compuesta o no de múltiples subunidades[6]
Cuando se ha identificado y purificado una nueva proteína, surgen inmediatamente dos preguntas:
- ¿Existe la proteína en condiciones fisiológicas como una cadena polipeptídica única o está formada por múltiples subunidades (multímero)?
- Si la proteína funcional tiene más de una subunidad, ¿son las subunidades idénticas o las hay de varios tipos?
 Se trata de una electroforesis en gel SDS en la que se observa, a la izquierda, un patrón de bandas modelo, y el patrón de badas de tres proteínas más
Se trata de una electroforesis en gel SDS en la que se observa, a la izquierda, un patrón de bandas modelo, y el patrón de badas de tres proteínas másDeterminación del número y peso aproximado de las subunidades: electroforesis en gel con SDS
Una vez se ha determinado el peso molecular nativo de la proteína, mediante la técnica del equilibrio de sedimentación, la manera más fácil de averiguar el peso o pesos moleculares de las subunidades consiste en utilizar la electroforesis en gel en presencia de SDS.
En estas condiciones, en presencia del SDS, la estructura secundaria, terciaria y cuaternaria de las proteínas se descomponen todas. La cadena se despliega y se rodea de moléculas SDS. Las numerosas cargas negativas transportadas por las muchas moléculas de SDS unidas a la proteína hacen que la carga que transporta la proteína sea insignificante, quedando la proteína cargada negativamente. La cadena polipeptídica plegada se transforma en un objeto alargado, cuya carga es proporcional a la longitud (y por tanto, al peso molecular) de la cadena. Así pues, no hay ninguna proteína carada positivamente después de pasar por un baño en SDS, y además la cantidad de carga negativa será independiente de la secuencia de aminoácidos, ya que únicamente dependerá de la longitud de la proteína.
Estas partículas (proteínas cargadas negativamente por efecto del SDS) migrarán en la electroforesis en gel con movilidades relativas, que dependen únicamente de sus longitudes. Entonces, si la electroforesis de la cadena proteica desconocida se lleva a cabo en el mismo gel que un conjunto de estándares de referencia, el peso molecular de la cadena desconocida puede medirse por interpolación en un gráfico. De esta manera conseguimos que las proteínas se separen por peso molecular gracias al efecto de un campo eléctrico que las separa por su cantidad de carga, pero como la carga depende únicamente de la longtud y el peso molecular de la proteína, éstas quedan separadas según su peso molecular
Cuando se investigan las subunidades de una proteína mediante esta técnica, es aconsejable realizar dos experimentos: uno en presencia de un agente reductor de enlaces disulfuro como el β-mercaptoetanol y otro en su ausencia. De este modo, se distinguirá entre las subunidades que se mantienen unidas mediante puentes disulfuro y las que se mantienen juntas sólo mediante fuerzas no covalentes.[6] [5]
Conclusiones
- Si se encuentra una sola banda en cada uno de estos geles con SDS, correspondiente en peso molecular a la de la proteína nativa, podemos concluir que la proteína existe en condiciones fisiológicas como una única cadena polipeptídica.
- Si la banda o bandas observadas son de un peso molecular muy inferior, significa que se trata de un multímero (proteína con múltiples subunidades).
Determinación del tipo de subunidades de la proteína multimérica: multímeros homotípicos o heterotípicos
Se muestran patrones de bandas en dos dimensiones. En la vertical según el punto isoeléctrico de las proteínas, y en horizontal, según su peso molecular. Esto permite diferenciar las distintas subunidades de los multímeros.
Suponiendo que el examen indique que hay varias subunidades, ¿hay sólo un tipo o hay varios? La presencia de más de una banda en el gel con SDS es una indicación clara de que hay más de una subunidad. Pero hallar una única banda no demuestra que las subunidades sean idénticas. Porque puede haber varios tipos de subunidades con secuencias de aminoácidos distintas, pero con pesos moleculares idénticos; estas subunidades distintas no pueden diferenciarse con gel SDS. Para dejar claro que sólo está presente un tipo de cadena, se debe recurrir a otros métodos. Por ejemplo, el enfoque isoeléctrico (asilando las subunidades sin detergentes o agentes cariotrópicos) puede ser una técnica muy sensible.
Por lo tanto, hay que aislar estas subunidades y hacer un examen sin modificar su carga para poder así hallar diferentes subunidades en un mismo patrón de bandas en la electroforesis.
Un método muy utilizado en el laboratorio es la electroforesis bidimensional o Western Blot, que consiste en separar las proteínas primero según su punto isoeléctrico, y después según su peso molecular. De esta forma podemos obtener información sobre el tipo de subunidades que presentan las proteínas multiméricas.
En la electroforesis 2D se obtiene una imagen como la que se observa a la derecha.
Determinación de los pesos moleculares exactos de las subunidades de las proteínas: espectrometría de masas

En los últimos años (2000-actual) se ha aplicado una técnica física antigua, la espectrometría de masas, para el estudio de las proteínas, que ha dado muy buenos resultados.
En el espectrómetro de masas, las moléculas se ionizan y posteriormente se aceleran a través del vacío mediante un campo eléctrico. Las partículas con distinta relación masa/carga se separan mediante su deflexión en un campo magnético o bien midiendo simplemente su tiempo de vuelo hasta un detector.
Cada molécula recibe una carga determinada (normalmente sólo unas pocas unidades electrónicas, + o -)durante su ionizació. El campo eléctrico contiene un ánodo (la fuente) y un cátodo, que contiene una pequeña apertura para que pasen las moléculas. El campo eléctrico proporciona a cada molécula una energía cinética proporcional a su carga.
Consideremos dos macromoléculas de masa diferente, cada una de las cuales recibe una unidad de carga positiva. La energía cinética para cada una será la misma, pero sus velocidades serán diferentes, ya que E=1/2m(v^2) (fórmula de la energía cinética). Esto significa que, cuanto mayor sea la molécula, se moverá más lentamente.
De esta forma, midiendo el tiempo de llegada al detector a través de la región de deriva, en el espectrómetro de masas, se puede medir la masa molecular.
Los resultados pueden ser muy exactos, del orden de una parte por 10.000 para moléculas del tamaño de la hemoglobina, lo cual es suficientemente exacto para detectar pequeños cambios de la secuencia de aminoácidos.[6] [5]
La técnica se ha ampliado para poder estudiar moléculas del tamaño de las inmunoglobulinas (alrededor de 150.000 Da).Bibliografía
- ↑ a b c d David L. Nelson, Michael M. Cox, Claudi M.Cuchillo; Lehninger - Principios de Bioquímica; cuarta edición(2005); editorial Omega; ISBN: 84-282-1410-7
- ↑ a b c d e f g h Trudy McKee, James R. McKee; Bioquímica - La base molecular de la vida; tercera edición (2003); editorial McGraw Hill; ISBN: 84-486-0524-1
- ↑ a b c d Werner MÜller-Esterl; Bioquímica - Fundamentos para Medicina y Ciencias de la Vida; primera edición (2008); editorial Reverté; ISBN: 978-84-291-7393-2
- ↑ a b c d e f g Christopher K. Mathews, K. E. van Holde,Kevin G. Ahern; Bioquímica, tercera edición (2002); editorial Addison Wesley; ISBN: 84-7829-053-2
- ↑ a b c David L. Nelson, Michael M. Cox, Claudi M.Cuchillo; Lehninger - Principios de Bioquímica; cuarta edición (2005); editorial Omega; ISBN: 84-282-1410-7
- ↑ a b c Christopher K. Mathews, K. E. van Holde,Kevin G. Ahern; Bioquímica, tercera edición (2002); editorial Addison Wesley; ISBN: 84-7829-053-2; páginas 234 y 235
Enlaces externos
- Proteopedia website La enciclopedia de proteínas en 3D y otras moléculas.
- PQS server
- 3D complex Clasificación estructural de complejos proteicos.
- PDB (Banco de Datos sobre Proteínas - Protein Data Bank)
Wikimedia foundation. 2010.
