- Galactoquinasa
-
Galactoquinasa 1[1] 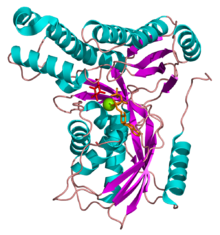
Figura 1. Modelo de cintas de un monómero de la galactoquinasa 1 humana unida a la galactosa (en rojo) y a un análogo del ATP (en naranja). La esfera verde representa un ion de magnesio.Otros nombres Galactosa quinasa HUGO 4118 Símbolo GALK1 Datos genéticos Código de gen GALK1 Tipo de gen Gen codificante Locus Cr. 17 q23-q25 Estructura/Función proteica Tamaño 392 (aminoácidos) Bases de datos Número EC 2.7.1.6 Entrez 2584 OMIM 604313 RefSeq NM_000154 UniProt P51570 N-acetilgalactosamina kinasa[2] Otros nombres Galactosaquinasa 2 HUGO 4119 Símbolo GALK2 Datos genéticos Código de gen GALK2 Tipo de gen Gen codificante Locus Cr. 15 [1] Estructura/Función proteica Tamaño 458 (aminoácidos) Bases de datos Entrez 2585 OMIM 137028 RefSeq NM_002044 UniProt Q01415 La galactoquinasa (EC 2.7.1.6) es una enzima fosfotransferasa que cataliza la fosforilación de la D-galactosa a D-galactosa-1-fosfato utilizando ATP como donante del grupo fosfato.[3]
- D-galactosa + ATP
 α-D-galactosa-1-fosfato + ADP
α-D-galactosa-1-fosfato + ADP
La galactoquinasa cataliza la primera etapa de la ruta de Leloir, una ruta metabólica encontrada en muchos organismos para el catabolismo de la β-D-galactosa a glucosa-1-fosfato.[4] La galactoquinasa que fue aislada por primera vez del hígado de mamíferos, ha sido estudiada extensivamente en las levaduras,[5] [6] archaeas,[7] plantas[8] [9] y humanos.[10] [11]
Contenido
Isozimas
En el ser humano existen dos genes que codifican galactoquinasas.
- El gen GALK1 codifica la galactoquinasa 1.
- El gen GALK2 codifica la N-acetilgalactosamina kinasa. Esta enzima actúa como una galactoquinasa cuando la galactosa está presente en altas concentraciones. De hecho se la conoce también como galactoquinasa 2.[12]
Estructura
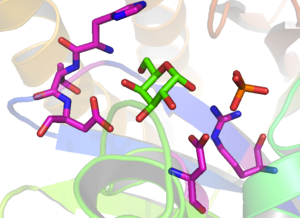 Figura 2. Estructura cristalina del sitio activo de la galactoquinasa del Lactococcus lactis.[13] La galactosa se muestra en verde, el fosfato en naranja y los residuos responsables de la unión del azúcar a la enzina en magenta: Arg-36, Glu-42, Asp-45, Asp-183 y Tyr-233. Arg-36 y Asp-183 de la galactoquinasa del Lactococcus lactis son análogos a los residuos Arg-37 y Asp-186 de la galactoquinasa humana.(From PDB 1PIE)
Figura 2. Estructura cristalina del sitio activo de la galactoquinasa del Lactococcus lactis.[13] La galactosa se muestra en verde, el fosfato en naranja y los residuos responsables de la unión del azúcar a la enzina en magenta: Arg-36, Glu-42, Asp-45, Asp-183 y Tyr-233. Arg-36 y Asp-183 de la galactoquinasa del Lactococcus lactis son análogos a los residuos Arg-37 y Asp-186 de la galactoquinasa humana.(From PDB 1PIE)
La galactoquinasa está compuesta por dos dominios separados por un gran hueco. Las dos regiones son conocidas como los dominios N- y C-terminal. El anillo adenina del ATP se une en un agujero hidrofóbico localizado en la interfaz entre los dos dominios. El dominio N-terminal está compuesto por 5 filamentos formados de láminas beta y 5 hélices alfa. El dominio C-terminal está caracterizado por dos capas de láminas beta antiparalelas y 6 hélices alfa.[10] La galactoquinasa no pertenece a la familia de las quinasas que actúan sobre carbohidratos, pero sí a la superfamilia de enzimas dependientes del ATP conocida como superfamilia GHMP quinasas.[14] GHMP es una abreviatura referente a los miembros originales de la familia: galactoquinasa, homoserina kinasa, mevalonato kinasa y fosfomevalonato kinasa. Los miembros pertenecientes a la superfamilia GHMP tienen una gran similitud en su estructura tridimensional aunque solamente comparten entre un 10% y un 20% de su secuencia. Estas enzimas contienen tres motivos bien conservados (I, II y III), el segundo participa en la unión del nucleótido y tiene la secuencia Pro-X-X-X-Gly-Leu-X-Ser-Ser-Ala.[13]
Especificidad
En diferentes especies, las galactoquinasas presentan una gran variedad de especificidad de sustrato.
- La galactoquinasa de la Escherichia coli puede fosforilar 2-deoxi-D-galactosa, 2-amino-deoxi-D-galactosa, 3-deoxi-D-galactosa y fucosa. La enzima no puede tolerar ninguna modificación en el carbono 4 (C-4) de la galactosa, pero los cambios en el C-2 no interfieren con la función de la enzima.[15]
- Las galactoquinasas de los humanos y ratas también son capaces de fosforilar la 2-deoxi-D-galactosa.[16] [17]
- Contrariamente la galactoquinasa de la Saccharomyces cerevisiae es altamente específica por la D-galactosa y no puede fosforilar glucosa, manosa, arabinosa, fucosa, lactosa, galactitol o 2-deoxi-D-galactosa.[5] [6]
Por otra parte, las propiedades cinéticas de la galactoquinasa también difieren en las diferentes especies.[10]
Mecanismo
Recientemente se han aclarado los roles de los aminoácidos del sitio activo de la galactoquinasa humana. El Asp-186 extrae un protón del C1-OH de la α-D-galactosa y el alcóxido nucleófilo resultante ataca el γ-fosfato del ATP. Un grupo fosfato es transferido al azúcar y el Asp-186 es deprotonado por agua. El residuo cercano Arg-37 estabiliza a Asp-186 en su forma aniónica y se ha provado que es esencial para la función de la galactoquinasa mediante experimentos de mutaciones puntuales.[11] Los residuos Asp y Arg del sitio activo están altamente conservado en las galactoquinasas.[10]
 Figura 3. Mecanismo de la galactoquinasa.[11] El residuo aspartato es estabilizado en su forma aniónica por un residuo arginina cercano.
Figura 3. Mecanismo de la galactoquinasa.[11] El residuo aspartato es estabilizado en su forma aniónica por un residuo arginina cercano.
Función biológica
La ruta de Leloir cataliza la conversión de galactosa en glucosa. La galactosa se encuentra en los productos lácteos, frutas y vegetales, y puede ser producida de forma endógena por la rotura de las glicoproteínas y glicolípidos. En la ruta de Leloir participan tres enzimas: galactoquinasa, galactosa-1-fosfato uridiltransferasa y UDP-galactosa 4-epimerasa. La galactoquinasa cataliza la primera etapa del metabolismo de la galactosa formando galactosa-1-fosfato.[4] [18]
Relevancia clínica
La galactosemia es un desorden metabólico raro caracterizado por la capacidad reducida de metabolizar galactosa y puede ser producido por una mutación en cualquiera de las tres enzimas de la ruta de Leloir.[4] La deficiencia en galactoquinasa, también conocida como galactosemia tipo II, es un desorden metabólico recesivo causado por una mutación en la galactoquinasa humana. Alrededor de unas 20 mutaciones que causan galactosemia tipo II han sido identificadas. El principal síntoma es la aparición de cataratas a una edad temprana. En las células del cristalino del ojo humano, la aldehído reductasa convierte galactosa a galactitol. Como la galactosa no es catabolizada a glucosa debido a una mutación en la galactoquinasa, el galactitol se acumula. El gradiente de galactitol a través de la membrana de las células del cristalino provoca que entre agua en las células provocando que se hinchen y una eventual apoptosis de las células.[19] El tratamiento más efectivo para la galactosemia es eliminar la lactosa de la dieta.
Enlaces externos
Referencias
- ↑ «GALK1». Consultado el 20 de noviembre de 2011.
- ↑ «GALK2». Consultado el 20 de noviembre de 2011.
- ↑ «ENZYME entry: EC 2.7.1.6». Consultado el 20 de noviembre de 2011.
- ↑ a b c Frey PA (Mar 1996). «The Leloir pathway: a mechanistic imperative for three enzymes to change the stereochemical configuration of a single carbon in galactose». FASEB J 10 (4): pp. 461–70. PMID 8647345.
- ↑ a b Schell MA, Wilson DB (May 1979). «Purification of galactokinase mRNA from Saccharomyces cerevisiae by indirect immunoprecipitation». J Biol Chem 254 (9): pp. 3531–6. PMID 107173.
- ↑ a b Sellick CA, Reece RJ (Jun 2006). «Contribution of Amino Acid Side Chains to Sugar Binding Specificity in a Galactokinase, Gal1p, and a TranscriptionalInducer, Gal3p». J Biol Chem 281 (25): pp. 17150–5. doi:. PMID 16603548.
- ↑ Hartley A, Glynn SE, Barynin V, Baker PJ, et al (Mar 2004). «Substrate specificity and mechanism from the structure of Pyrococcus furiosus galactokinase». J Mol Biol 337 (2): pp. 387–98. doi:. PMID 15003454.
- ↑ Foglietti MJ, Percheron F (1976). «Purification and mechanism of action of a plant galactokinase». Biochimie 58 (5): pp. 499–504. PMID 182286.
- ↑ Dey PM (Oct 1983). «Galactokinase of Vicia faba seeds». Eur J Biochem 136 (1): pp. 155–9. doi:. PMID 6617655.
- ↑ a b c d Holden HM, Thoden JB, Timson DJ, Reece RJ (Oct 2004). «Galactokinase: structure, function and role in type II galactosemia». Cell Mol Life Sci 61 (19–20): pp. 2471–84. doi:. PMID 15526155.
- ↑ a b c Megarity CF, Huang M, Warnock C, Timson DJ (Mar 2011). «The role of the active site residues in human galactokinase: Implications for the mechanisms of GHMP kinases». Cell Mol Life Sci 39 (3): pp. 120–126. doi:.
- ↑ «N-acetylgalactosamine kinase». Consultado el 20 de noviembre de 2011.
- ↑ a b Thoden JB, Holden HM (Aug 2003). «Molecular structure of galactokinase». J Biol Chem 278 (35): pp. 33305–11. doi:. PMID 12796487.
- ↑ Tang M, Wierenga K, Elsas LJ, Lai K (Dec 2010). «Molecular and biochemical characterization of human galactokinase and its small molecule inhibitors». Chem Biol Interact 188 (3): pp. 376–85. doi:. PMID 20696150.
- ↑ Yang J, Fu X, Jia Q, Shen J, et al (Jun 2003). «Studies on the substrate specificity of Escherichia coli galactokinase». Org Lett 5 (13): pp. 2223–6. doi:. PMID 12816414.
- ↑ Timson DJ, Reece RJ (Nov 2003). «Sugar recognition by human galactokinase». BMC Biochem 4: pp. 16. doi:. PMID 14596685.
- ↑ Walker DG, Khan HH (Jun 1968). «Some properties of galactokinase in developing rat liver». Biochem J 108 (2): pp. 169–75. PMID 5665881.
- ↑ Holden HM, Rayment I, Thoden JB (Nov 2003). «Structure and function of enzymes of the Leloir pathway for galactose metabolism». J Biol Chem 278 (45): pp. 43885–8. doi:. PMID 12923184.
- ↑ Timson DJ, Reece RJ (Apr 2003). «Functional analysis of disease-causing mutations in human galactokinase». Eur J Biochem 270 (8): pp. 1767–74. doi:. PMID 12694189.
Categorías:- Genes del cromosoma 17
- Genes del cromosoma 15
- Transferasas
- D-galactosa + ATP
Wikimedia foundation. 2010.